Indholdsfortegnelse:
- Trin 1: Opret Adjacency Matrix
- Trin 2: Opret relationer
- Trin 3: Tilføj sygdomsstatistik
- Trin 4: Randomiser chancen for, at en vaccineret og ikke -vaccineret person kan blive inficeret
- Trin 5: Opret matricer for mennesker, der er uvaccinerede og inficerede ud fra første oplysninger
- Trin 6: Plot indledende graf
- Trin 7: Simuler infektionens progression
- Trin 8: Brug Monte Carlo -teorien
- Trin 9: Lav filen ('infectSim.m') med simuleringen i en funktion
- Trin 10: Beregn procentdelen af ikke -vaccinerede og vaccinerede mennesker, der blev inficeret
- Trin 11: Opret en outputvariabel i din funktion 'infectSim.m'
- Trin 12: Opret en menu for at få de første betingelser for simuleringen fra brugeren
- Trin 13: Vælg en procentdel af uvaccinerede mennesker og beregne gennemsnittet af uvaccinerede og inficerede for den valgte procentdel
- Trin 14: Graf: 'Tendensen til infektion i uvaccinerede vs. Vaccineret mod specificeret sygdom '
- Trin 15: Slutprodukt: Sådan ser simuleringen ud
Video: At blive vaccineret eller ej? et projekt om at observere besætningens immunitet gennem sygdomsimulering: 15 trin

2024 Forfatter: John Day | [email protected]. Sidst ændret: 2024-01-30 08:29
Oversigt over projektet:
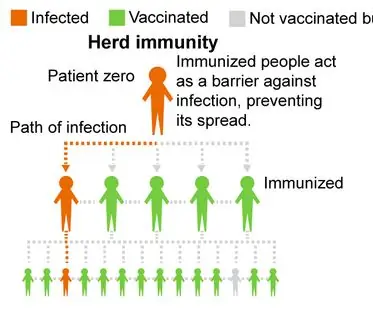
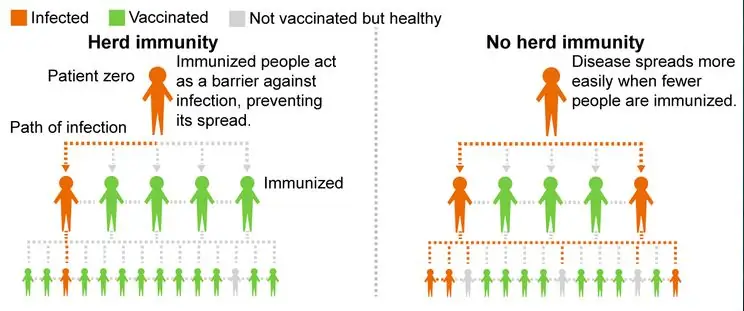
Vores projekt undersøger besætningsimmunitet og håber at tilskynde folk til at få vaccinationer for at reducere infektionshastigheden i vores lokalsamfund. Vores program simulerer, hvordan en sygdom inficerer en befolkning med forskellige procentdele af vaccinerede og uvaccinerede satser. Det viser besætningsimmunitet ved at vise, hvordan et øget antal af den vaccinerede befolkning kan reducere antallet af berørte mennesker.
Vi modellerer dette i Matlab ved hjælp af grafteoretiske begreber. Grafteori er en matematisk måde at repræsentere forholdet mellem objekter. I grafteorien har grafer hjørner (eller noder) forbundet med kanter (eller linjer). For vores projekt er knudepunkterne de berørte personer, og kanterne er deres forbindelser. For eksempel, hvis to noder er forbundet med en kant, betyder det, at de er "venner" eller har en eller anden form for kontakt med hinanden. Denne kontakt er en måde for sygdommen at sprede sig på. Det er derfor, vi brugte grafteori til at modellere vores koncept, fordi vi ville se, hvordan sygdom spredes blandt individer, der er forbundet i en befolkning.
Vores projekt involverer også Monte Carlo -metoden. Monte Carlo -metoden er algoritmer, der skaber gentagne stikprøver for at modtage numeriske resultater. I vores projekt bruger vi denne metode til at køre vores simulering flere gange og ændre procentdelen af de første uvaccinerede for at se, hvor hurtigt folk bliver smittet.
Al projektkode er knyttet nederst!
PC-kredit:
Matlab-link til grafteori:
Trin 1: Opret Adjacency Matrix
Opret et nyt script. Vi vil kalde vores 'infektionSim.m'.
Vi skal oprette en variabel 'NUMOFPEOPLE'. Du kan tildele den til en hvilken som helst heltalværdi. Dette repræsenterer antallet af mennesker i din befolkning.
Fra nu af vil vi antage det
NUMOFPEOPLE = 20;
Start først med at bruge Matlabs grafteorifunktioner til en ikke -styret graf.
Hvis du er interesseret i at lære mere, er her et link, hvor du kan læse mere i dybden.
www.mathworks.com/help/matlab/math/directed-and-undirected-graphs.html
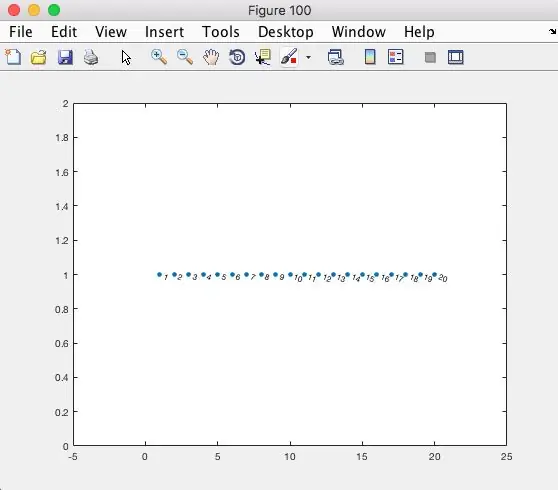
Oprettede en adjacency matrix.
adjMatrix = nuller (NUMOFPEOPLE);
Dette vil skabe en firkantet matrix på 0s. Hver række i matrixen er en person. Hver kolonne i matrixen er en person eller ven, som personen møder i løbet af dagen.
Se figur 100 (ovenfor) for at hjælpe med at visualisere, hvordan adjMatrix ser ud for 20 personer.
** Fra dette tidspunkt antager vi, at NUMOFPEOPLE er lig med 20. **
Du kan prøve at plotte denne tilstødelsesmatrix. Her er lidt mere information om afbildning af disse typer matricer.
Bemærk: Sådan fungerer adjacensmatrix.
Eks:
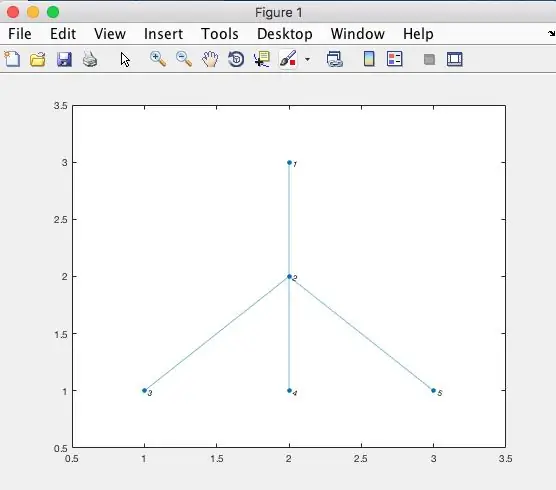
%at lave den tilstødende matrix
a = [0, 1, 0, 0, 0; 1, 0, 1, 1, 1; 0, 1, 0, 0, 0; 0, 1, 0, 0, 0; 0, 1, 0, 0, 0] %afbildning g = graf (a); %ved hjælp af graffunktionen (grafteori) figur (1); h = plot (g);
Se figur 1 (ovenfor) for at se, hvordan du tilføjer kanter i tilpasningsmatrixen, ved hjælp af koden i "Bemærk".
Trin 2: Opret relationer
Nu hvor personerne (hjørner eller noder) er oprettet, skal vi oprette et netværk af relationer (linjer eller kanter på grafen). Dette vil simulere, hvordan mennesker interagerer og møder andre mennesker i løbet af en dag.
Dette kan gøres på mange måder. En måde at fuldføre denne opgave på er først at tildele et vilkårligt tal til hver person for at bestemme, hvor mange mennesker hver person vil interagere med på en dag.
numOfFriendsMatrix = randi ([leastFriendsPersonCanHave, mostFriendsPersonCanHave], 1, NUMOFPEOPLE);
Dette gør en 1 til 20 matrix af tilfældige heltal, der repræsenterer antallet af interaktioner, hver person har om dagen. Kolonnerne i denne matrix ville være det tal, der svarer til hver person. For eksempel, hvis vi tildeler minstFriendsPersonCanHave = 2 og mostFriendsPersonCanHave = 5, ville vi få tilfældige værdier mellem 2 og 5.
Har du problemer med randi ()? Skriv i terminalen
hjælp randi
Dernæst laver vi en randomiseret matrix (kaldet "allFriendsmatrix") af, hvordan hver person i befolkningen er forbundet/interagerer inden for befolkningen.
tempMatrix = ;
count = 0; allFriendsMatrix = ; for k = 1: NUMOFPEOPLE mens længde (tempMatrix) ~ = numOfFriendsMatrix (k) count = count +1; temp = randi ([1, NUMOFPEOPLE]); tempMatrix (count) = temp; slut rydde hver mens længde (tempMatrix) ~ = 9 tempMatrix = [tempMatrix, NaN]; afslut allFriendsMatrix = [allFriendsMatrix; tempMatrix]; tempMatrix = ; count = 0; ende
Dybdegående forklaring af kode:
Først opretter vi en tom midlertidig matrix til at indeholde hver persons venner/interaktionsliste. Vi initialiserer også tælling, som bare holder styr på, hvor den nye tilfældige forbindelse skal placeres i tempMatrix. For -loops kører 20 gange, så dette sker for hver enkelt person i befolkningen. Den første mens loop løber, indtil hver persons tempMatrix er den samme længde af det tilfældigt tildelte antal interaktioner. I denne sløjfe genereres et tilfældigt tal, der svarer til en person i befolkningen, og placeres i tempMatrix. Fordi længderne på hver af tempMatrixes er forskellige, havde vi brug for at oprette nogle NaN -værdier, så vi kan sammenkæde alle disse tempMaticies til én matrix ('allFriendsMatrix'). Den anden while loop løser dette problem ved at tilføje NaN'er til hver tempMatrix. Mens loop var indstillet til at køre 9 gange, fordi det er et tal større end 5, hvilket var den øvre grænse for venner, en person kan tildeles. Værdien '9' er variabel og kan/skal ændres, når 'mostFriendsPersonCanHave' er større end 9. De sidste tre linjer med kode (eksklusive slutningen) tilføjer tempMatrix til den næste række i 'allFriendsMatrix'. Derefter rydder det tempMatrix og tæller for den næste person.
Produktion
Sådan skal output se ud for det første løb gennem for -loop (før de sidste tre linjer).
tempMatrix = 16 8 17 16 13 NaN NaN NaN NaN
allFriendsMatrix =
16 8 17 16 13 NaN NaN NaN NaN 8 8 2 7 11 NaN NaN NaN NaN 10 13 NaN NaN NaN NaN NaN NaN NaN 11 17 2 NaN NaN NaN NaN NaN NaN 10 12 NaN NaN NaN NaN NaN NaN NaN NaN 4 13 2 12 NaN NaN NaN NaN NaN 17 10 9 3 1 NaN NaN NaN NaN 16 16 6 NaN NaN NaN NaN NaN NaN 3 8 17 17 14 NaN NaN NaN NaN 20 19 3 NaN NaN NaN NaN NaN NaN 13 10 NaN NaN NaN NaN NaN NaN NaN 2 18 10 16 NaN NaN NaN NaN NaN 2 6 14 3 13 NaN NaN NaN NaN 8 16 14 8 NaN NaN NaN NaN NaN 7 7 NaN NaN NaN NaN NaN NaN NaN 19 10 9 NaN NaN NaN NaN NaN NaN 10 19 NaN NaN NaN NaN NaN NaN NaN 5 18 NaN NaN NaN NaN NaN NaN NaN 1 7 NaN NaN NaN NaN NaN NaN NaN 16 7 13 10 1 NaN NaN NaN NaN
Tilføj derefter disse relationer til adjMatrix.
for hverRække = 1: NUMOFPEOPLE
for eachCol = 1: 9 hvis isnan (allFriendsMatrix (eachRow, eachCol)) == 0 adjMatrix (eachRow, allFriendsMatrix (eachRow, eachCol)) = 1; adjMatrix (allFriendsMatrix (eachRow, eachCol), eachRow) = 1; ende ende ende
Kode Forklaring
Denne dobbelte for loop går gennem hver række og kolonne i 'allFriendsMatrix'. If -sætningen kører for alle værdier, der ikke er 'NaN'. Grundlæggende vil det skabe kanterne eller linjerne i grafen. Så den første linje, dette vil gøre, er person 1 til person 16 og person 16 til person 1. Fordi den ikke er styret, skal 1 ændres for begge! Vi kan ikke bare have kanten 1 til 16 og ikke 16 til 1. De skal være symmetriske for at den kan køre korrekt i Matlab.
I vores simulering fastslog vi, at mennesker ikke kan interagere med sig selv. Når vi randomiserede værdierne, er der en chance for, at vores tilstødende matrix har disse fejl.
Lad os rette dette med følgende kode:
for hver = 1: NUMOFPEOPLE
adjMatrix (hver, hver) = 0; ende
Kode Forklaring
Denne for loop sikrer, at person 1 ikke er forbundet til person 1, person 2 ikke er forbundet til person 2 osv. Ved at gøre dem alle 0. Som du kan se nedenfor i output -sektionen, har vi kvadratens diagonal matrix fra øverst til venstre til nederst til højre er alle 0'er.
Produktion
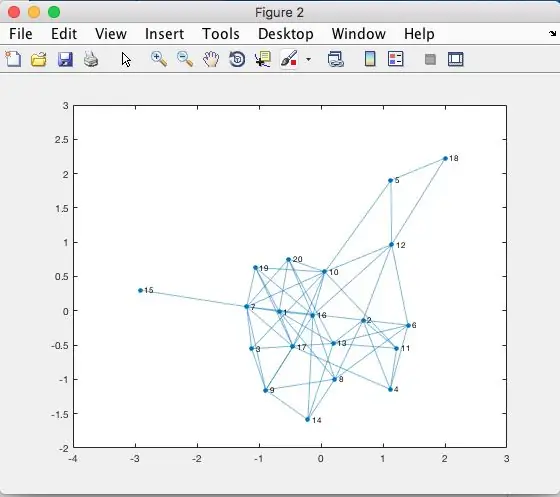
Dette er den sidste adjMatrix for denne aktuelle simulering. Dette tegner sig for alle linjerne i grafen (figur 2).
adjMatrix =
0 0 0 0 0 0 1 1 0 0 0 0 1 0 0 1 1 0 1 1 0 0 0 1 0 1 1 1 0 0 1 1 1 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 1 0 0 1 0 0 0 0 0 0 0 0 1 0 0 0 1 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 0 0 0 0 0 1 0 0 0 1 0 1 0 0 0 1 0 0 0 1 1 0 0 0 0 0 0 0 1 1 1 0 0 0 0 0 1 1 0 0 0 0 1 0 1 0 1 1 1 1 0 0 0 1 0 0 1 0 0 0 0 1 0 1 0 0 0 0 0 0 1 0 0 0 1 1 0 0 0 0 0 1 0 1 1 0 0 0 0 0 1 0 1 0 1 0 0 0 1 1 0 0 0 1 1 0 1 1 0 1 0 1 0 0 0 0 0 1 0 0 1 0 0 0 0 0 0 0 0 1 0 0 1 1 0 0 0 1 0 0 0 0 0 1 0 1 0 0 1 1 1 0 0 1 0 0 0 0 1 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 0 1 1 0 0 0 1 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 1 1 1 0 1 0 1 0 0 0 0 1 1 1 0 0 1 0 0 1 0 1 1 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 1 0 0 0 0 0 1 1 0 0 0 1 0 0 0 0 0 1 0 0 1 0 0 1 0 0 1 0 0 0 0
Se figur 2 for at se grafen over 'adjMatrix'.
Trin 3: Tilføj sygdomsstatistik
Nu hvor dit program kan oprette en graf med et sæt tilfældige mennesker og oprette tilfældige forhold, skal vi indtaste sygdomens oplysninger eller statistik for at se, hvordan disse interaktioner i en befolkning kan øge eller reducere infektionen.
Opret disse variabler:
unvacc %type: dobbelt; procent chance for, at uvaccinerede ikke får sygdommen
vacc %-type: dobbelt; procent chance for, at vaccinerede ikke får sygdommen unvacc_perc %type: double; procent befolkning uvaccineret init_infect %type: int; procent befolkning vaccineret
Dernæst skal vi lave nogle beregninger.
Vi skal lave en 'infectMat', som er en 3*NUMOFPEOPLE matrix.
vacc_perc = 1-unvacc_perc;
infectMat = nan (3, NUMOFPEOPLE); nummer = rund (vacc_perc * NUMOFPEOPLE); infektionMat (1, 1: tal) = vacc; infektionMat (1, nummer+1: slut) = unvacc; infektionMat (2, 1: ende) = 0; infektionMat (2, 1: init_infect) = 1;
Kode Forklaring
linje 1: Procentdel af befolkningen uvaccineret beregnet
linje 2: opret en 3*N antal personer matrix
linje 3: find ud af antallet af personer, der er vaccineret fra vaccineret procentdel
linje 4: for de vaccinerede mennesker, giv dem en immunitet forbundet med at have vaccinen. Denne værdi tildeles baseret på forskning om sygdommen.
linje 5: giv dem procentvis immunitet for resten af befolkningen (uvaccinerede personer). Denne værdi tildeles baseret på forskning om sygdommen.
linje 6: indstil oprindeligt alle mennesker til ikke at blive inficeret.
linje 7: For antallet af personer, der oprindeligt blev inficeret, skal du udfylde de første par kolonner i overensstemmelse hermed.
Nu hvor vi har angivet alle parametrene for sygdomssimuleringen, vil vi randomisere chancen for, om personen (både vaccineret og ikke -vaccineret) bliver inficeret. Dette gøres i det næste trin ved at tildele tilfældige værdier mellem 0 og 1 til hver person i tredje række af denne 'infectMat'.
Trin 4: Randomiser chancen for, at en vaccineret og ikke -vaccineret person kan blive inficeret
Derefter skal du tildele hver person et tilfældigt nummer, dette vil blive brugt senere til at afgøre, om personen bliver inficeret eller ej.
for w = 1: længde (infectMat)
infektionMat (3, w) = rand; ende
Kode forklaring
Denne for loop omhandler den tredje række af 'infectMat', der blev oprettet i det sidste trin. 'rand' tildeler en værdi mellem 0 og 1 til hvert indeks i række 3.
Produktion
infectMat er nu færdig! Dette var med en befolkning med 100% vaccination og 1 person oprindeligt inficeret.
infektionMat =
Kolonner 1 til 12 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 1,0000 0 0 0 0 0 0 0 0 0 0 0 0,0869 0,5489 0,3177 0,9927 0,7236 0,5721 0,7172 0,9766 0,4270 0,9130 0,8973 0,8352 Kolonner 13 til 0,0075 0,7500 0,7500 0 0 0 0 0 0 0 0 0,0480 0,3593 0,2958 0,6291 0,1336 0,3740 0,8648 0,2503
række 1: Procentvis chance for IKKE at få sygdommen
række 2: inficeret eller ikke inficeret (boolsk værdi)
række 3: Nummer, der bruges til at kontrollere, om en person, der ikke er inficeret, bliver smittet, hvis de møder en inficeret person. Hvis en inficeret person møder en inficeret person, er dette tal større end tallet i række 1 (for samme kolonne), så er de inficeret. Vi koder denne funktionalitet i trin 7.
Trin 5: Opret matricer for mennesker, der er uvaccinerede og inficerede ud fra første oplysninger
Opret 2 matricer kaldet "matrixUnvacc" og "matrixInfected", der gemmer alle de inficerede personer fra infektionMat. Dette vil blive brugt, så vi kan farvekode grafen for dem, der er inficerede, uvaccinerede eller vaccinerede, og hjælper med at visualisere virkningen af uvaccinerede versus vaccinerede personer.
rydde hver
matrixInfected = ; matrixUnvacc = ; for h = 1: længde (infektionMat) hvis infektionsMat (1, h) == unvacc matrixUnvacc = [matrixUnvacc, h]; ende ende for person = 1: NUMOFPEOPLE hvis infektionsMat (2, person) == 1 matrixInfected = [matrixInfected, person]; ende ende
Kode Forklaring
Opret to tomme matricer for at gemme numrene på de mennesker, der er henholdsvis uvaccinerede og inficerede. Både for loops køres 20 gange, og hvis if -sætningen er tilfreds, tilføjes tallet til den korrekte matrix.
Produktion
matrixUnvacc =
matrixInfected =
[1]
Trin 6: Plot indledende graf
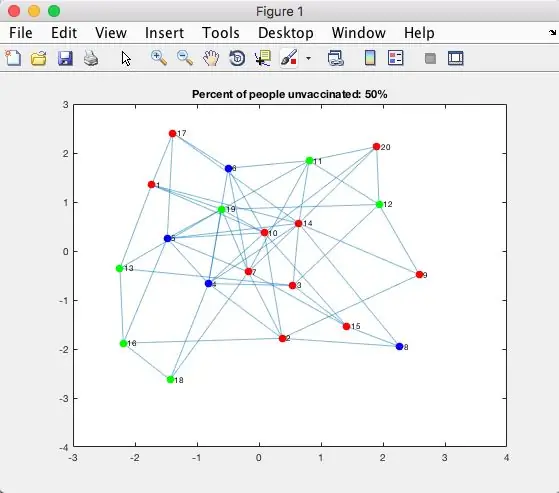
Dernæst skal vi plotte adjacensmatrixen.
g = graf (adjMatrix);
figur (1) p = plot (g, 'NodeColor', 'b', 'MarkerSize', 7); fremhæve (p, matrixUnvacc, 'NodeColor', 'g') fremhæve (p, matrixInfected, 'NodeColor', 'r') title_unvacc = unvacc_perc*100; title (['Procent af personer uvaccinerede:', num2str (title_unvacc), '%']); pause (hastighed)
Kode forklaring
Grafteori i Matlab har indbygget funktioner. Når vi bruger grafen () -funktionen, er vi i stand til at oversætte 'adjMatrix' til en egentlig uorienteret graf. Vi skal derefter oprette et plot ved hjælp af funktionen plot () for faktisk at se, hvordan det ser ud. Vi indstiller dette plot () til en variabel, så vi lettere kan manipulere og ændre plottets farver under hele simuleringen. Alle mennesker (eller noder) er indledningsvis indstillet til farven 'blå'. Derefter indstilles alle de uvaccinerede mennesker til farven 'grøn'. De inficerede personer sættes derefter til farven 'rød'. Titlen er indstillet i henhold til den procentvise værdi af uvaccinerede mennesker, der testes. Funktionen pause () stopper midlertidigt MatLab -udførelsen. Vi passerer den variable hastighed, som spredes, som beregnes i sekunder.
Se billedet (ovenfor) for at se en tilfældig farvekodet graf.
Lær mere om funktionen highlight () i MatLab.
Trin 7: Simuler infektionens progression
Dernæst skal vi finde ud af, hvem der bliver inficeret efter interaktionerne (registreret i adjMatrix) og opdatere grafen, når nogen bliver inficeret.
Brug adjMatrix til at bestemme, hvilke mennesker der er inficeret efter deres interaktion med mennesker på en dag.
for hverRække = 1: længde (adjMatrix)
hvis infectMat (2, eachRow) == 1 for eachCol = 1: length (adjMatrix) if adjMatrix (eachRow, eachCol) == 1 % eachRow = personen % eachCol = dens ven % hver persons ven og se om de bliver inficeret. hvis infectMat (3, eachCol)> infektionMat (1, eachCol) infektionMat (2, eachCol) = 1; fremhæve (p, eachCol, 'NodeColor', 'r') pause (hastighed) ende ende ende ende ende
For -loop sløjfer gennem hver person. Den kontrollerer, at hvis personen er inficeret, vil den kontrollere hver enkelt person/ven, de interagerede med, og kontrollere, om venens immunitetsniveau var større end sygdommens styrke. Det er her, 'infectMat', vi skabte tidligere, spiller ind. 1. og 3. række i hver vens kolonne sammenlignes, og hvis 3. række er større, betyder det, at vennen ikke havde en høj nok immunitet til at undslippe sygdommen og i sidste ende bliver inficeret. Vi skifter også til farve ved hjælp af fremhævning () til rød, hvis de bliver inficeret.
Nu skulle din kode til simuleringen fungere! og for enhver befolkningsstørrelse skal du bare ændre NUMOFPEOPLE!
Trin 8: Brug Monte Carlo -teorien
For at tage dette et skridt videre og udtrække data fra vores simulator ('infectSim.m'), ønskede vi at beregne og tegne tendensen i procent af uvaccinerede mennesker, der blev inficeret, og procentdelen af vaccinerede mennesker, der blev smittet. Vi antager, at procentdelen af vaccinerede mennesker, der blev smittet, skulle være meget lavere end procentdelen af uvaccinerede, der blev smittet.
Trin 9: Lav filen ('infectSim.m') med simuleringen i en funktion
For at køre Monte Carlo ville vi gerne køre simuleringen flere gange og akkumulere data, så vi kan bruge det til at tegne procentdelen af mennesker, der blev inficeret.
Funktionen kan sættes op således:
funktionsoutput = infectSim (unvacc, vacc, NUMOFPEOPLE, unvacc_perc, init_infect, speed)
Kommenter variablerne i din simulering, da du nu sender disse ind gennem hovedfilen (vi begynder at skrive dette i trin 12):
unvacc, vacc, NUMOFPEOPLE, unvacc_perc, init_infect
Den nye variabel
hastighed
vil blive tildelt i hovedfilen (Monte_Carlo.m).
Bemærk: Glem ikke slutningen i bunden af funktionsfilen for at afslutte funktionen!
Trin 10: Beregn procentdelen af ikke -vaccinerede og vaccinerede mennesker, der blev inficeret
Dette beregner procentdelen af uvaccinerede mennesker, der blev smittet. Denne kode findes i bunden af filen 'infectSim.m'.
number_of_unvacc = 0;
number_of_infec_unvacc = 0; %beregner procentdel af uvaccinerede mennesker, der blev inficeret for x = 1: length (infektionMat), hvis infektionMat (1, x) == unvacc nummer_of_unvacc = nummer_of_unvacc+1; afslutte, hvis infektionMat (1, x) == unvacc & infektionMat (2, x) == 1 nummer_of_infec_unvacc = nummer_of_infec_unvacc +1; slut slut percent_of_unvacc_and_infec = (number_of_infec_unvacc / number_of_unvacc)*100;
Kode forklaring
I for -løkken vil den løkke over NUMOFPEOPLE gange. Hver gang antallet i infektionMat svarer til unvacc -nummeret (dvs. 0,95 == 0,95), vil antallet af uvaccinerede personer blive øget med 1. Hver gang antallet i infektionsmaten svarer til unvacc -nummeret, og de er inficeret, vil antallet af inficerede og uvaccinerede stiger med 1. Den sidste linje dividerer antallet af inficerede, uvaccinerede mennesker med det samlede antal uvaccinerede. Derefter beregnes procentdelen ud fra dette.
Udfordring:
Prøv at beregne procentdelen af vaccinerede af mennesker, der blev smittet! (Tip: det ligner meget ovenstående kode, men nogle af variablerne ændres og navne justeres.)
Derefter beregnes procentdelen af de inficerede mennesker baseret på den samlede befolkning:
pre_per_infect = cumsum (infectMat (2,:));
per_infect = (pre_per_infect (1, NUMOFPEOPLE)/NUMOFPEOPLE)*100;
Kode forklaring
Den kumulative sum beregnes ved hjælp af den anden række af infektionsMat, som gemmer 1'er og 0'er afhængigt af, om personen er inficeret eller ej. Da funktionen cumsum () giver en matrix tilbage, tager vi den sidste værdi i matrixen ('pre_per_infect (1, NUMOFPEOPLE)'), som skal være den faktiske sum af alle værdierne fra 'infectMat (2,:)'. Ved at dividere summen med NUMOFPEOPLE og gange det med 100 får vi den sidste procentdel af inficerede i den samlede befolkning.
Trin 11: Opret en outputvariabel i din funktion 'infectSim.m'
output = [per_infect, percentage_of_unvacc_and_infec, percentage_of_vacc_and_infec];
Kode forklaring
Gem disse oplysninger i output, som sendes tilbage til main (Monte_Carlo.m), når funktionen kaldes og er færdig med at køre. Disse data bruges til at tegne point for procent af de smittede blandt dem, der er vaccinerede og ikke -vaccinerede.
Din 'infectSim.m' -funktion skal udføres nu! Det vil dog ikke køre, fordi vi stadig mangler at skrive main!
Trin 12: Opret en menu for at få de første betingelser for simuleringen fra brugeren
Husk, hvordan vi sagde variablen
hastighed
ville blive oprettet og passeret gennem hovedfunktionen? Vi er nødt til at opnå værdierne for at videregive til funktionen. Bemærk, rækkefølgen af værdierne, når du kalder funktionen, betyder noget!
Start med at bede brugeren om at skrive nogle svar i terminalen.
> Vælg en sygdom. Bemærk, at det er store og små bogstaver >> Kighoste >> Influenza >> Mæslinger >> Udvalgt sygdom: Influenza >> Vælg populationsstørrelse. >> 20 >> 200 >> Valgt befolkning: 20 >> Vælg simuleringshastighed. >> Hurtig >> Langsom >> Valgt hastighed: Hurtig
Denne kode nedenfor spørger brugeren, hvilken sygdom de vil undersøge.
disp ('Vælg en sygdom. Bemærk, at det er følsomt over for store og små bogstaver')
fprintf ('Kighoste / nFlu / nMeasles / n') sygdom = input ('Sygdom valgt:', 's'); hvis ulige (sygdom, 'kighoste') vacc =.85; %15 procent chance for at få sygdom unvacc =.20; %80 procent chance for at få sygdom, hvis ikke ulighed (sygdom, 'influenza') vacc =.75; %25 procent chance for at få sygdom unvacc =.31; %69 procent chance for at få sygdom, hvis ikke ulighed (sygdom, 'Mæslinger') vacc =.97; %3 procent chance for at få sygdom unvacc =.10; %90 procent chance for at få sygdom slut
Kode forklaring:
Funktionen disp () udskriver erklæringen til skærmen, og den udskriver også de forskellige muligheder. Sygdommen vil blive tildelt i overensstemmelse hermed. Denne version er i øjeblikket ikke ansvarlig for ugyldigt input. Ugyldigt input vil producere en fejl og stoppe programmet helt. Hver sygdom har vacc og unvacc -værdier forbundet med det. Disse værdier er IKKE tilfældige. Vi fik disse værdier ved at undersøge statistik om sygdommene.
Dernæst skal vi spørge brugeren, om de vil teste en stor eller lille befolkningsstørrelse for deres valgte sygdom.
disp ('Vælg befolkningens størrelse.')
fprintf ('20 / n200 / n ') speed = input (' Befolkning valgt: ',' s '); hvis isequal (hastighed, '20') population_størrelse = 20; elseif isequal (hastighed, '200') population_størrelse = 200; ende
Kode forklaring
Dette udskriver en erklæring til brugeren og beder brugeren om at indtaste, hvilken befolkningsstørrelse den vil teste. Denne version er i øjeblikket ikke ansvarlig for ugyldigt input. Ugyldigt input vil producere en fejl og stoppe programmet helt. 20 blev plukket, fordi det er en lille stikprøvestørrelse, der stadig giver en god idé om, hvordan infektion spredes i en lille population. 200 mennesker blev valgt som den større mulighed, fordi 200 punkter afbildet på grafen næsten ikke havde nogen overlapning af punkter, så alt let kunne ses og skelnes fra hinanden.
Dernæst skal vi finde simuleringens hastighed.
disp ('Vælg simuleringshastighed.')
fprintf ('Hurtig / nSlow / n') hastighed = input ('Valgt hastighed:', 's'); hvis isequal (hastighed, 'Hurtig') sim_speed = 0; elseif isequal (hastighed, 'Slow') sim_speed = 0,25; ende
Kode forklaring
Denne proces var den samme som at få typen af sygdom og befolkningens størrelse. For hurtigt vil der ikke være nogen pause. og for langsom vil der være en 0,25 sekunders forsinkelse i for -løkken, når simuleringen køres.
Store! Nu har vi alle input fra den bruger, vi har brug for! Lad os gå videre til at indsamle data for forskellige procentdele af uvaccinerede mennesker.
Trin 13: Vælg en procentdel af uvaccinerede mennesker og beregne gennemsnittet af uvaccinerede og inficerede for den valgte procentdel
Denne kode er for 0% af uvaccinerede mennesker.
% ------- % 0 Uvaccineret ------------
per_infect_av_0 = ; percentage_of_unvacc_and_infec_av_0 = ; for i = 1:20 ud = infektionSim (unvacc, vacc, population_size, 0, 1, sim_speed); per_infect_av_0 = [per_infect_av_0, out (1, 1)]; percent_of_unvacc_and_infec_av_0 = [percentage_of_unvacc_and_infec_av_0, out (1, 2)]; slut gennemsnittet_inficeret_0 = middelværdi (pr. infektion_av_0); average_unvacc_and_infected_0 = middelværdi (percentage_of_unvacc_and_infec_av_0);
Kode forklaring:
For -løkken køres 20 gange. Outputtet fra funktionen, infectSim (), gemmes ude. Hver gang for -loop løber, tilføjes procentdelen af inficerede i den samlede population til matrixen 'per_infect_av_0'. Derudover tilføjes også procentdelen af uvaccinerede og inficerede hver gang til matrixen 'percentage_of_unvacc_and_infec_av_0'. I de sidste to linjer beregnes disse to, ovennævnte matricer derefter i gennemsnit og lagres i variabler. For at opsummere, gemmes procentsatserne for hver simulering, beregnes i gennemsnit og tegnes grafisk. Monte Carlo bruges til at vise gennemsnitsværdien for at køre en simulering og vise resultatet. Til vores eksperimentelle formål vælger vi at køre simuleringen 20 gange og gennemsnit disse værdier.
Udfordring:
Gentag for alle procenter, du vil teste! Dette kan gøres ved at ændre variabelnavnene i henhold til procenttalene. Vi testede for 0%, 5%, 10%, 20%, 30%og 50%.
Antydning:
Den eneste linje, der skal ændres i den faktiske kode er
out = infectSim (unvacc, vacc, population_size, 0, 1, sim_speed);
Skift nul til procent i decimalform. For eksempel, for 5% uvaccineret simulering, bør 0'en erstattes med 0,5.
Trin 14: Graf: 'Tendensen til infektion i uvaccinerede vs. Vaccineret mod specificeret sygdom '
Dette er koden til at lave en graf over tendensen til infektion hos uvaccinerede personer mod uvaccinerede personer.
graph_mat_y = [average_infected_0, average_infected_5, average_infected_10, average_infected_20, average_infected_30, average_infected_50];
graf_mat_x = [0, 5, 10, 20, 30, 50]; hældning = (gennemsnitlig_inficeret_5-gennemsnitlig_infektion_0)/5; line_y = [gennemsnit_inficeret_0, (hældning*50)+gennemsnitlig_infektion_0]; line_x = [0, 50]; figur (2) plot (graf_mat_x, graf_mat_y); line (line_x, line_y, 'Color', 'red', 'LineStyle', '-'); title (['Trend i uvaccination for', sygdom]); xlabel ('Procentdel af initial uvaccineret'); ylabel ('Procentdel af sidste inficerede')
Kode forklaring
linje 1: tildelte y -værdier til gennemsnit af procent inficerede
linje 2: tildelt x -værdier til procentdelen af initialprocenten uvaccineret
linje 3: beregne hældningen på 0% og 5%
linje 4: gemme y -værdier for linje. Dette er en fortsættelse af sektionen 0% til 5%.
linje 5: gemme y -værdier for linje. Denne linje spænder over grafens længde.
linje 6: opret figur
linje 7: afbild grafen x og y -værdier for de procent inficerede, som er uvaccinerede.
linje 8: plot linjen. Dette bruges til at vise, at det ikke stiger lineært, men eksponentielt.
linje 9: Indstil titel til grafen.
linje 10-11: Angiv x- og y-etiketter til graf.
Nu skal du kunne se, at jo større procentdel af befolkningen ikke er vaccineret, jo større infektionsmængder. Du vil også se, at de fleste af de prikker, der bliver røde, er grønne prikker, hvilket viser, at vaccinen i nogen grad hjælper! Håber du kunne lide denne vejledning. Kommenter, hvis du har spørgsmål!
Trin 15: Slutprodukt: Sådan ser simuleringen ud
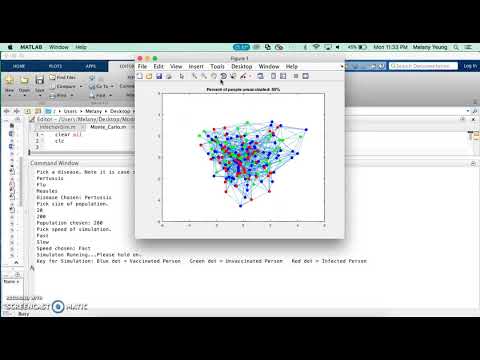
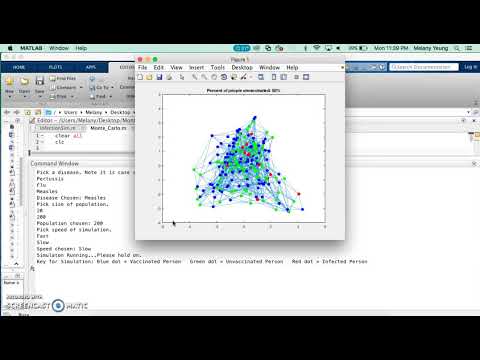
Al koden kan findes her
Anbefalede:
Lodning gennem hulkomponenter - Grundlaget for lodning: 8 trin (med billeder)

Lodning gennem hulkomponenter | Grundlaget for lodning: I denne instruktionsbog vil jeg diskutere nogle grundlæggende om lodning af gennemgående huller til kredsløb. Jeg går ud fra, at du allerede har tjekket de første 2 instruktioner til min Lodning Basics -serie. Hvis du ikke har tjekket mit In
Styr husholdningsapparater gennem Alexa med ESP8266 eller ESP32: 8 trin

Styr husholdningsapparater gennem Alexa med ESP8266 eller ESP32: Hey, hvad sker der, fyre! Akarsh her fra CETech.Dette projekt af mit vil hjælpe dit liv med at blive let, og du kommer til at føle dig som en konge efter at have kontrolleret apparater i dit hus ved blot at give en kommando til Alexa.Hovedsagen bag denne side
Gå online uden at blive snooped: Tor (The Onion Router): 4 trin (med billeder)

Gå online uden at blive snoket: Tor (The Onion Router): Når du går online, efterlader du spor overalt. Du kan hænge ud med venner på IM, tjekke websteder eller downloade musik. Hvis du bor i et land, hvor snokere nysgerrig efter, hvad almindelige borgere gør online (lke, um, U
Sådan lytter du til dit Itunes -bibliotek gennem din tv eller AV -modtager: 5 trin

Sådan lytter du til dit Itunes -bibliotek gennem din tv eller AV -modtager: Okay, jeg gjorde dette instruerbart på 10 minutter. Det var så let! En bærbar computer ville være god til dette, men et skrivebord i nærheden af et tv er også okay. Hvis du har en Airport Express, vil jeg tilføje et trin til jer mennesker. (Bemærk: Jeg ejer ikke en lufthavnstransport, så hvis jeg
Sådan får du dit TV-B-til at blive usynligt : 6 trin (med billeder)

Sådan får du dit TV-B-til at blive usynligt …: Hej, tak for din interesse. Først og fremmest undskylder jeg enhver sprogfejl, jeg kunne gøre i denne vejledning, jeg er fransk (ingen er perfekte; p) Fik fri til at fortælle mig, om der er nogle rettelser, thx;) Nu er det væsentlige: Efter at have brugt min TV-B
