Indholdsfortegnelse:
- Trin 1: Trin 1: Indlæsning af billede
- Trin 2: Trin 2: Støjfiltrering og histogram
- Trin 3: Trin 3: Indstilling af tærskler
- Trin 4: Oprettelse af GUI
- Trin 5: Videodemo
Video: MatLab Lungesegmentering: 5 trin

2024 Forfatter: John Day | [email protected]. Sidst ændret: 2024-01-30 08:25
Af: Phuc Lam, Paul Yeung, Eric Reyes
Anerkender, at fejl i lunger segmentering vil producere falske oplysninger om identifikation af et sygdomsområde og kan direkte påvirke diagnoseprocessen. Moderne computerstøtte teknikker kunne ikke levere nøjagtige resultater, når lungesygdomme har udfordrende former. Disse unormale former kan være forårsaget af pleural effusioner, konsolideringer osv. Ved anvendelse af lunge -segmenteringsteknikken, hvor lungens grænser er isoleret fra omgivende thoraxvæv, kan vores app identificere grænserne med brugerens input -tærskler for at give fuldt tilpassede visninger af lungernes former, Formålet med dette MatLab-projekt er at oprette en brugervenlig interaktiv lungesegmenteringsapp til at detektere patologiske forhold i lungernes røntgenbilleder. Vores mål er at skabe en mere effektiv måde at illustrere og identificere unormale lunger for at give læger og radiologer en mere pålidelig måde at diagnosticere lungesygdomme på. Ved hjælp af appdesignværktøjet i MatLab er programmet designet til at arbejde specifikt med røntgenbilleder af brystet og computertomografi (CT), men det er også testet til at arbejde med MR-scanninger.
Instruktionerne herunder indeholder vores støjfiltreringsteknik (lavpas Wiener-filter) samt billedgrænsen (ved hjælp af intensitetshistogrammet i gråtonebilledet) og ved hjælp af en morfologisk gradient (forskellen mellem udvidelse og erosion af et billede) til identificere en interesseområde. Instruktionen forklarer derefter, hvordan vi integrerer alle elementerne i den grafiske brugergrænseflade (GUI).
Bemærk:
1). Dette projekt er inspireret af en forskningsartikel: "Segmentering og billedanalyse af unormale lunger ved CT: aktuelle tilgange, udfordringer og fremtidige tendenser". Som kan findes her
2). Vi bruger røntgenbilleder fra NIH: Clinical Center. Link kan findes her
3). Hjælp til appdesignere kan findes her
4). Inden koden køres: skal du ændre Dir -stien (i linje 34) til din filmappe og billedtype (linje 35) (vi analyserer *.png).
Trin 1: Trin 1: Indlæsning af billede
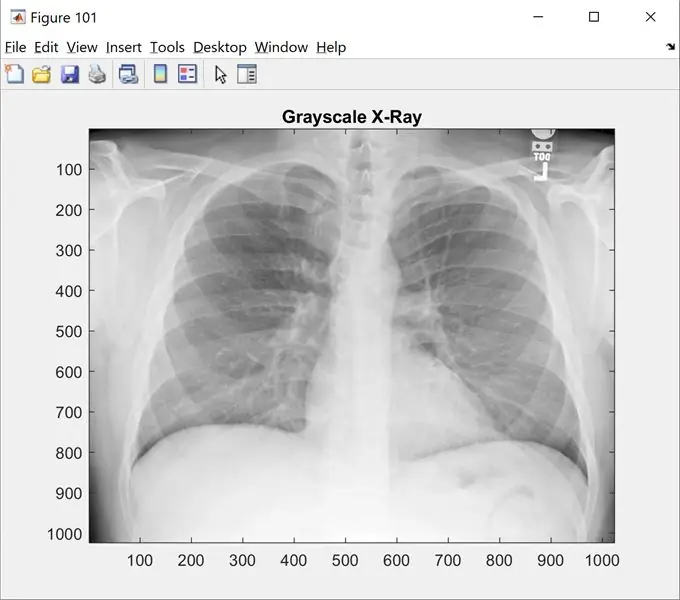
Dette trin viser dig det originale billede i gråtoner. Skift 'name_of_picture.png' til dit billednavn
klar; clc; luk alle;
%% Indlæser billeder
raw_x_ray = 'name_of_picture.png';
I = imread (raw_x_ray);
figur (101);
imshow (I);
farvekort (grå);
titel ('gråtonerøntgen');
Trin 2: Trin 2: Støjfiltrering og histogram
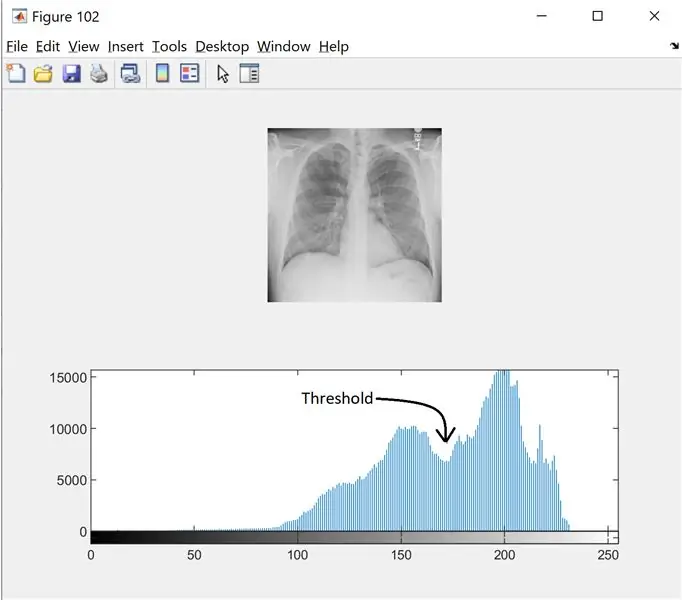
For at finde tærsklen for gråtonebilledet, ser vi på histogrammet for at se, om der er forskellige tilstande. Læs mere her
I = wiener2 (I, [5 5]);
figur (102);
delplot (2, 1, 1);
imshow (I);
delplot (2, 1, 2);
imhist (I, 256);
Trin 3: Trin 3: Indstilling af tærskler
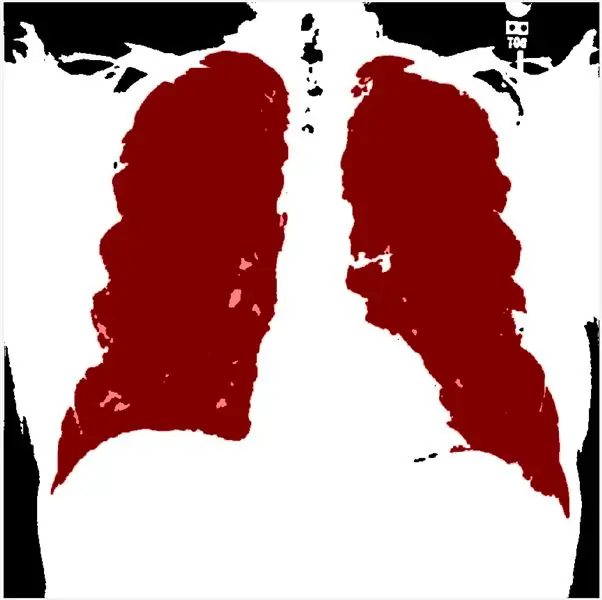
Dette trin giver dig mulighed for at indstille tærsklen i henhold til histogrammet. morfologiskGradient vil fremhæve området med interesse for rødt, og funktionen grænseoverskridende overlejrer det skitserede og filtrerede billede af lungen i rødt.
Ved at bruge regionprops kan vi præcisere arrays af soliditet og sortere dem i faldende. Dernæst binariserer jeg det grå sclae -billede og anvender den morflogiske gradientmetode og mLoren Shurasking for at fremhæve interesseområdet (ROI). Det næste trin er at vende billedet, så lunge -ROI'et er hvidt i den sorte baggrund. Jeg bruger funktionen showMaskAsOverlay til at vise 2 masker. Bemærk: koden er inspireret af Loren Shure, link.
Lasly, jeg opretter rød kontur ved at bruge bwbwgrænserne og maskere filterbilledet og grænserne.
a_thresh = I> = 172; % angiver denne tærskel
[labelImage, numberOfBlobs] = bwlabel (a_thresh);
rekvisitter = regionprops (a_thresh, 'alle');
sortedSolidity = sort ([props. Solidity], 'descend');
SB = sortedSolidity (1);
hvis SB == 1 % SB kun accepterer soliditet == 1 filtrer knogler ud
binaryImage = imbinarize (I); figur (103);
imshow (binaryImage); farvekort (grå);
SE = strel ('square', 3);
morphologicalGradient = imsubtract (imdilate (binaryImage, SE), imerode (binaryImage, SE));
mask = imbinarize (morphologicalGradient, 0,03);
SE = strel ('square', 2);
mask = tæt (maske, SE);
maske = imfill (maske, 'huller');
maske = bwareafilt (maske, 2); % kontrol antal områdeshow
notMask = ~ maske;
maske = maske | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
showMaskAsOverlay (0,5, maske, 'r'); % du skal downloade app/funktion showMaskAsOverlay
BW2 = imfill (binaryImage, 'huller');
nyt_billede = BW2;
ny_billede (~ maske) = 0; % inverter baggrund og huller
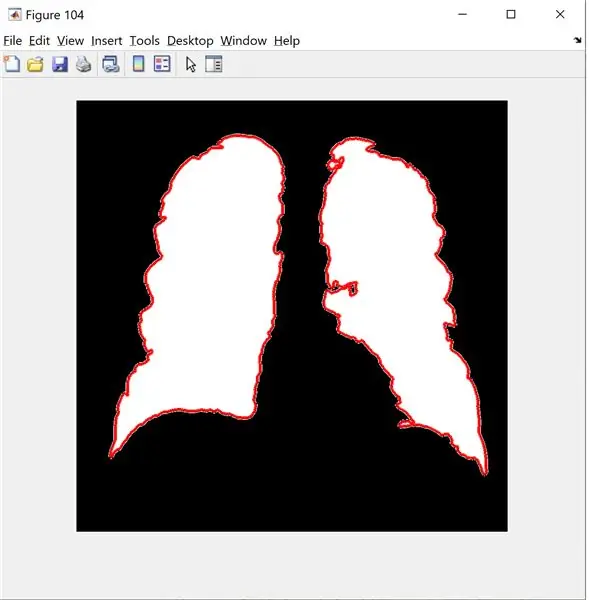
B = bwgrænser (ny_billede); % kan kun acceptere 2 dimensioner
figur (104);
imshow (nyt_billede);
hold fast
grænseoverskridende (B);
ende
Trin 4: Oprettelse af GUI
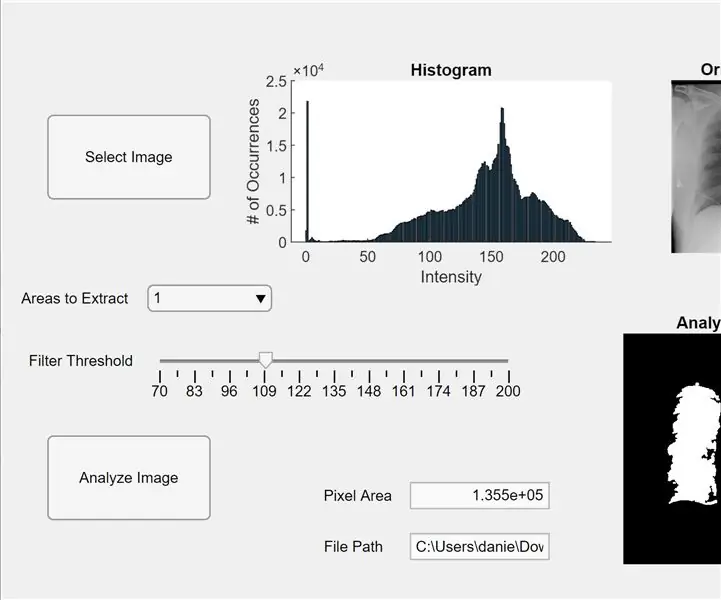
Nu integrerer vi den tidligere kode i en MATLAB -app. Åbn App Designer i MATLAB (Ny> App). Først designer vi grænsefladen ved at klikke-hold-og trække i tre akser ind i det centrale arbejdsområde. Derefter klikker vi på og holder to knapper, et redigeringsfelt (tekst), et redigeringsfelt (numerisk), en skyder og en rullemenu. To akser vil hver vise forhåndsvisningen og analysere billedet, og den tredje akse vil vise et histogram af pixels for det "valgte" eksempel. Redigeringsfeltet (tekst) -boksen viser filstien for det valgte billede, og redigeringsfeltet (numerisk) viser det detekterede pixelområde i lungerne.
Skift nu fra designvisning til kodevisning i App Designer. Indtast koden for ejendomme i koden ved at klikke på den røde "Egenskaber" -knap med et plustegn ved det. Initialiser egenskaberne I, tærskel og regionsToExtract som i koden nedenfor. Højreklik derefter på en knap i øverste højre side af arbejdsområdet (komponentbrowseren) og gå fra Tilbagekald> Gå til … tilbagekald. Tilføj koden til "funktion SelectImageButtonPushed (app, begivenhed)." Denne kode giver dig mulighed for at vælge et billede, der skal analyseres fra din computer ved hjælp af uigetfile. Efter valg af et billede vises et eksempelbillede under akserne ledsaget af et histogram. Højreklik derefter på den anden knap og gentag den samme procedure for at oprette en tilbagekaldsfunktion.
Tilføj koden under "funktion AnalyzeImageButtonPushed (app, begivenhed)." Denne kode udfører pixeltælling og blobdetektering på forhåndsvisningsbilledet på knappen Analysebillede (hvilken som helst du har højreklikket på for denne kode). Efter programmering af knapperne programmerer vi nu skyderen og rullemenuen. Højreklik på skyderen, opret en tilbagekaldsfunktion og tilføj koden under "funktion FilterThresholdSliderValueChanged (app, begivenhed)" indtil slutningen. Dette gør det muligt for skyderen at justere tærsklen for grå intensitet.
Opret en tilbagekaldsfunktion til rullemenuen, og tilføj koden under "funktion AreastoExtractDropDownValueChanged (app, begivenhed)" for at give rullemenuen mulighed for at ændre antallet af klatter, der vises på de analyserede billedakser. Klik nu på hver enhed i komponentbrowseren og ændr deres egenskaber efter din smag, f.eks. Ændring af navne på objekter, fjernelse af akser og ændring af skalering. Træk og slip enhederne i komponentbrowseren i designvisning til et funktionelt og letforståeligt layout. Du har nu en app i MATLAB, der kan analysere billeder af lunger for pixelområde!
ejendomme (Adgang = privat) I = ; % billedfil
tærskel = 257; %tærskel for binærisering af grå intensitet
regionToExtract = 2;
ende
funktion SelectImageButtonPushed (app, begivenhed)
clc; Dir = 'C: / Users / danie / Downloads / images_004 / images'; %definer variabel fil "præfiks"
[imageExt, sti] = uigetfile ('*. png'); %tag den variable del af billednavnet
imageName = [Dir filesep imageExt]; %sammenkædede uændrede og variable spændinger
app. I = imread (imageName); %læste billedet
imshow (app. I, 'parent', app. UIAxes); %viser billedet
app. FilePathEditField. Value = sti; %visningsfilsti for, hvor det originale billede kom fra
ende
function AnalyzeImageButtonPushed (app, begivenhed)
originalImage = app. I;
originalImage = wiener2 (app. I, [5 5]); %prikfjernelsesfilter
histogram (app. AxesHistogram, app. I, 256); %vis histogram for billede
a_thresh = originalImage> = app.threshold; % angiver denne tærskel
labelImage = bwlabel (a_thresh);
rekvisitter = regionprops (a_thresh, 'alle');
sortedSolidity = sort ([props. Solidity], 'descend');
SB = sortedSolidity (1);
hvis SB == 1 % SB kun accepterer soliditet == 1 filtrer knogler ud
SE = strel ('square', 3);
morphologicalGradient = imsubtract (imdilate (labelImage, SE), imerode (labelImage, SE));
mask = imbinarize (morphologicalGradient, 0,03);
SE = strel ('square', 2);
mask = tæt (maske, SE);
maske = imfill (maske, 'huller');
mask = bwareafilt (mask, app.regionsToExtract);
% kontrol antal områdeshow
notMask = ~ maske;
maske = maske | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
BW2 = imfill (labelImage, 'huller');
nyt_billede = BW2;
ny_billede (~ maske) = 0;
B = bwgrænser (nyt_billede); % kan kun acceptere 2 dimensioner imshow (new_image, 'parent', app. UIAxes2);
hold (app. UIAxes2, 'on');
grænseoverskridende (B);
sæt (gca, 'YDir', 'reverse');
lungArea = bwarea (new_image);
app. PixelAreaEditField. Value = lungArea;
ende
ende
funktion FilterThresholdSliderValueChanged (app, begivenhed)
app.threshold = app. FilterThresholdSlider. Value;
ende
function AreastoExtractDropDownValueChanged (app, event) stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double (stringNumber);
ende
ende
Anbefalede:
MATLAB Let ansigtsregistrering: 4 trin

MATLAB Easy Face Detection: Hovedformålet med disse instruktører er at vise, hvor let billedbehandlingen vil være.Med hjælp af MATLABFace -registrering og -sporing har været et vigtigt og aktivt forskningsfelt, så derfor vil jeg forklare hvordan kan det gøres med
Sådan kodes og udgives Matlab 2016b til Word (Begyndervejledning): 4 trin

Sådan kodes og udgives Matlab 2016b til Word (Beginners Guide): Matlab er et højtydende sprogprogram, der bruges til at beregne tekniske resultater. Det har evnen til at integrere visuals, beregninger og programmering på en brugervenlig måde. Med dette program kan brugeren offentliggøre problemer og løsninger
Styring af Raspberry Pi ved hjælp af Matlab: 5 trin

Styring af Raspberry Pi ved hjælp af Matlab: Hey, denne vejledning handler om at kontrollere din hovedløse hindbærpi ved hjælp af matlab. Du skal muligvis installere matlab til den nyeste version for at understøtte et nyere hindbær pi -bord
Matlab-baseret ROS Robotic Controller: 9 trin
Matlab-baseret ROS Robotic Controller: Lige siden jeg var barn har jeg altid drømt om at være Iron Man og gør det stadig. Iron Man er en af de karakterer, der er realistisk muligt og ganske enkelt håber jeg på at blive Iron Man en dag, selvom folk griner af mig eller siger, at det er umuligt
Forbedringer af togprogrammering i MATLAB: 6 trin

Forbedringer af togprogrammering i MATLAB: Mål: Målet med dette programmerede system er at se på en Arduino i lille skala og anvende kodningen i større skala for potentielt at forbedre visse sikkerhedsfunktioner ved Amtrak Railroad -systemer. For at gøre dette har vi tilføjet en jordfugtighedssen
