Indholdsfortegnelse:
- Trin 1: Start grafisk brugergrænseflade (GUI)
- Trin 2: Indlæsning og læsning af MR -billeder i MATLAB
- Trin 3: Billedfiltrering
- Trin 4: Tumorisolering gennem elliptisk maske
- Trin 5: Tumoropstilling
- Trin 6: Analyse af tumors fysiske egenskaber
Video: Hjernetumor MR -detektion ved hjælp af Matlab: 6 trin

2024 Forfatter: John Day | [email protected]. Sidst ændret: 2024-01-30 08:28
Af: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Denne MATLAB -kode er et program til at detektere den nøjagtige størrelse, form og placering af en tumor fundet i en patients hjernens MR -scanninger. Dette program er designet til oprindeligt at arbejde med tumordetektion i hjernens MR -scanninger, men det kan også også bruges til kræftdiagnostik i andre organscanninger.
De følgende instruktioner beskriver først metoderne til billedanalyse gennem filtrering og oprydning af MR -scanningen, gennem binærisering, median filtrering og glidende vinduer. Dernæst vil den instruere om, hvordan man isolerer tumoren ved hjælp af en på forhånd genereret elliptisk maske og filtrerer den yderligere for at skitsere omkredsen af tumorens form.
Når tumoren er opdaget, vil instruktionerne yderligere beskrive, hvordan dette program kan inkorporeres i en grafisk brugergrænseflade (GUI). Gennem disse instruktioner vil den relevante kode og filer blive vedhæftet for at forklare, hvordan denne MR -scanningsanalyse fungerer.
Nogle ting at vide, downloade og have klar, før du fortsætter med denne instruktive: 1. Sørg for at have den nyeste version af MATLAB downloadet. Du kan installere R2018b her:
2. For at køre dette program skal du have adgang til MR -hjernescanningsfiler. Selvom nogle altid kan findes fra Google -billeder, kan der udføres grundig og præcis analyse ud fra korrekte billeder af forskellige lag af hjernescanninger for hver patient. Du kan få adgang til filerne for 20 forskellige patienter med glioblastom før og efter behandling fra denne database:
3. Fokus for dette program og de forskellige metoder, der guider dette projekt, er skitseret i denne forskningsartikel:
Trin 1: Start grafisk brugergrænseflade (GUI)
Det første trin ville være at oprette og starte den grafiske brugergrænseflade, GUI. Dette kan gøres ved at skrive vejledning i kommandovinduet, trykke på enter og oprette en ny GUI. Når dette trin er fuldført, kan du begynde at oprette funktioner såsom akser, statisk tekst, redigere tekst og trykknapper, der vises, når programmet køres, og brugeren kan interagere med. Disse funktioner kan redigeres og manipuleres via ejendomsinspektøren, men den vigtigste funktion, der skal ændres, når du opretter disse funktioner, er tagnavnet. Det er vigtigt at ændre tagnavnet på hver funktion, der implementeres, fordi det giver os mulighed for at oprette en genkendelig tilbagekaldsfunktion. Når du er tilfreds med layoutet på din GUI, kan du fortsætte med at indlæse de DICOM -filer, der vises i GUI'en.
Trin 2: Indlæsning og læsning af MR -billeder i MATLAB
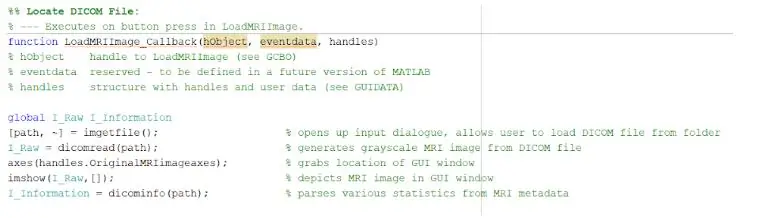

For at indlæse DICOM -filer skal du korrekt initialisere tilbagekaldsfunktionen, der ville blive udført, når du trykker på knappen "Indlæs MRI -billede." Når dette er fuldført, skal du oprette en global variabel, der viser billedet på håndtagets akser, hvor du vil have det originale MRI -billede vist. De MR -scanningsbilleder, der downloades fra databasen, er alle DICOM -formaterede filer, der skal indlæses i dit MATLAB -bibliotek. Find filen ved hjælp af imgetfile for at indlæse dem i programmet. Billederne læses ved hjælp af den indbyggede MATLAB -funktion 'dicomread', og det første råbillede for hver fil er indlejret i de venstre GUI -akser ved hjælp af imshow.
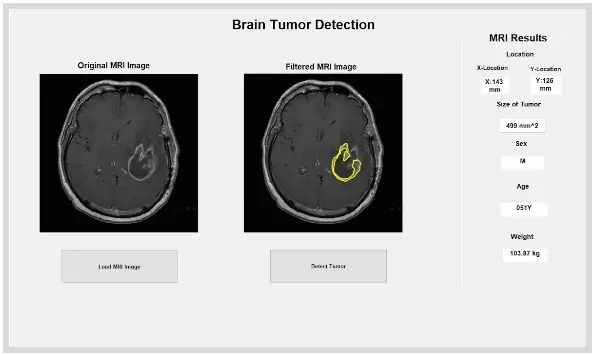
Den indbyggede MATLAB -funktion 'dicominfo' er også yderst nyttig til at adressere alle oplysninger om hver MRI dicom -fil. Vi brugte denne funktion til at udtrække alle beskrivende oplysninger om patienterne, såsom deres køn, alder, vægt og højde. Denne funktion giver dig også en stack -rækkefølge, som er nyttig til implementering af programmet inden for den grafiske brugergrænseflade. Vi skabte variabler for hver af de beskrivende oplysninger om patienterne, som vil blive brugt til GUI'en, når der trykkes på detekteringsknappen.
Trin 3: Billedfiltrering


Når DICOM -filen for det rå billede er blevet indlæst og læst, skal billedet konverteres fra gråtoner til en binær form, der kun består af sort -hvide pixels Vi brugte funktionen 'imbinarize' til at oprette et binært billede fra det rå billede ved at kontrollere aspekter af adaptiv tærskelværdi ved følsomhedsværdi på 0,59. Standard tærskelfølsomhedsfaktoren, 0,5 var lav og ude af stand til at registrere de lysere pletter og pletter fra billedet, så vi øgede den til 0,59.
Det binærede billede behandles derefter gennem et medianfilter ved hjælp af funktionen 'medfilt2', fordi det binære billede er todimensionalt. Vi indstiller hver outputpixel til at indeholde medianværdien i 5 x 5 -kvarteret omkring den tilsvarende pixel i inputbinariseret billede. Dette reducerer støj og bevarer kanterne i en 5 x 5 firkant omkring hver pixel. Derefter anvender vi et glidende vindue ved hjælp af 'strel' for at oprette et diskformet fladt strukturelement med en naboskabsradius på 2 for at identificere hver central, oprindelsespixel, i hvert diskområde. Vi brugte et diskstruktureringselement, fordi vi analyserer hvert cirkulært sted og pixelerne inden for hvert sted, så et diskformelement er mere nyttigt.
Når billedet er blevet filtreret, kan det rengøres ved hjælp af funktionen 'lukk' for at fjerne de sorte pletter mellem de filtrerede hvide pixels i billedet og lukke alle hullerne omkring det. Det fuldstændigt bearbejdede billede kan derefter plottes i den anden underplot af den forhåndstildelte figur, hvilket muliggør en sammenligning mellem det rå og filtrerede billede.
Trin 4: Tumorisolering gennem elliptisk maske



Tumorens lyspunkter kan derefter isoleres fra det hovedfiltrerede billede gennem en på forhånd genereret elliptisk maske. For at oprette denne maske skal du kende størrelsen på det originale, rå MRI-scanningsbillede, og ved hjælp af dets række- og kolonnelængde, som henholdsvis x og y- koordinater, allokere centerkoordinaterne til elliptisk. Vi indstiller y-aksen som en hovedakse med en radius på 50 enheder fra midten og den mindre akse med en radius på 40 enheder fra midten.
Vi brugte MATLAB-funktionen 'meshgrid' til at generere et kartesisk plan med todimensionale gitterkoordinater baseret på koordinaterne i vektorer fra 1 til længden af x-aksen og fra 1 til længden af y-aksen i billedet. Col er en matrix, hvor hver række er en kopi af x-aksen, og Row er en matrix, hvor hver kolonne er en kopi af y-aksen. Det kartesiske gitter repræsenteret af koordinaterne Col og Row har længder (1: Y_Size) rækker og længde (1: X_Size) kolonner. Brug indekserne for Col og Row genereret af det kartesiske gitter til at bestemme ellipsens ligning afhængigt af den forudbestemte radius og centerkoordinater. Den elliptiske kontur kan nu fyldes med de hvide pixels, der findes fra tumorpletter.
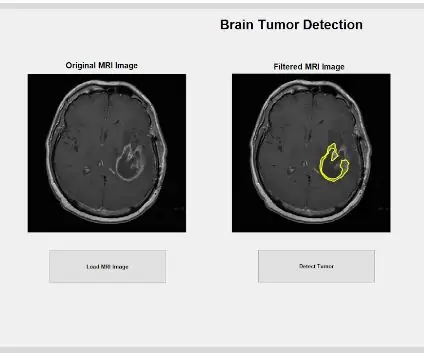
Ved hjælp af den på forhånd genererede elliptiske maske kan vi beskære den specifikke tumor, du ønsker at analysere, fra det filtrerede billede. Den elliptiske maske registrerer, hvilke pletter der logisk passer inden for ellipsens omrids, og accepterer dette som en plet på det filtrerede billede for at være acceptabel som en tumor. Funktionen 'bwareafilt' filtrerer derefter alle andre objekter uden for denne detekterede tumor fra billedet. Vi brugte et specifikt vindue på 500 x 4000 empirisk baseret på alle billedernes dimensioner. Vi påførte derefter et andet glidende vindue med 'strel' som et fladt skiveformet strukturelement med en større naboskabsradius på 6 for at lukke hullerne mellem hver central hvid pixel inden for den detekterede tumor. Den påviste tumorplet renses yderligere ved hjælp af 'imclose' for yderligere at fjerne de sorte pixels og udfylde alle hullerne med 'imfill'. Denne forarbejdede tumor kan derefter vises i den tredje delplot i det forudlokerede plot for at give en sammenligning mellem den isolerede tumor og de originale og filtrerede billeder af MR -scanningen.
Trin 5: Tumoropstilling
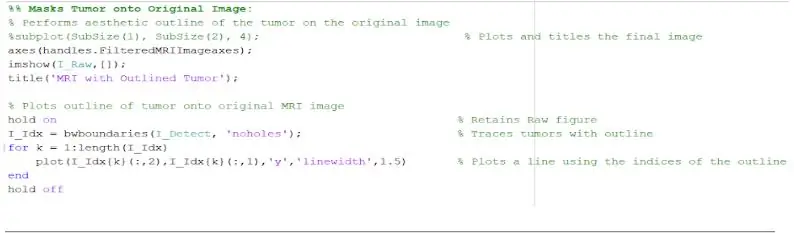

Nu hvor tumoren er isoleret med masken, kan den skitseres og vises på det originale billede for at vise dens nøjagtige placering. For at gøre dette brugte vi funktionen 'bwboundaries' til at spore den tidligere detekterede tumor med en kontur. Vi specificerede omridset til ikke at inkludere hullerne i tumorobjektet, som det skitseres. Dette kan afbildes på det originale, rå billede ved hjælp af en 'for' -sløjfe, der tegner omridset omkring tumoren ved hjælp af linjens indeks med en linjebredde på 1,5 pixels. Denne kontur er derefter afbildet på det rå billede, der viser den nøjagtige størrelse og placering af tumoren i forhold til den originale MR -scanning.
Trin 6: Analyse af tumors fysiske egenskaber
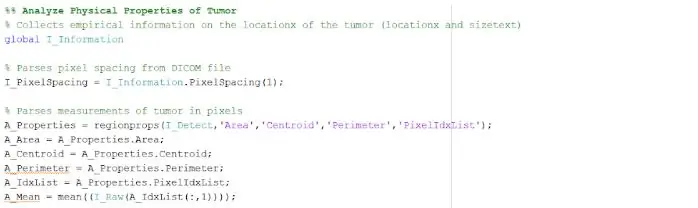
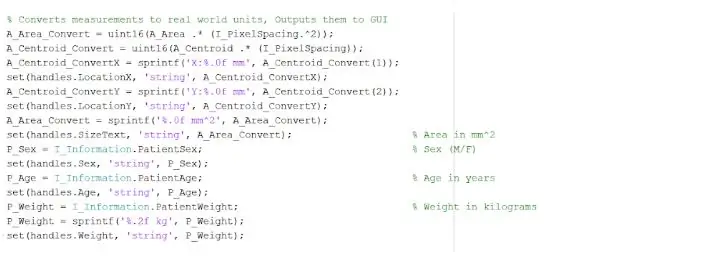
Det isolerede og skitserede sted kan give os nyttige oplysninger om tumorens størrelse, område og placering. Vi brugte funktionen 'regionprops' til at detektere tumorens egenskaber, der vedrører areal, omkreds, centroider og pixelindeksværdien. Denne pixelindeksværdi giver os enhederne i den virkelige verden for hver pixel i hvert billede, unikt for hver scanning. Disse egenskaber kan derefter konverteres til enheder i den virkelige verden på millimeter. De empiriske oplysninger, programmet giver os, er unikke for hver MR -scanning og er yderst nyttige til at bestemme størrelsen, placeringen og typen af tumor, som brugerne kan analysere og indarbejde i den grafiske brugergrænseflade.
Anbefalede:
DIY -- Sådan laver du en edderkoprobot, der kan kontrolleres ved hjælp af smartphone ved hjælp af Arduino Uno: 6 trin

DIY || Sådan laver du en edderkoprobot, der kan styres ved hjælp af smartphone Brug af Arduino Uno: Mens du laver en edderkoprobot, kan man lære så mange ting om robotik. Ligesom at lave robotter er underholdende såvel som udfordrende. I denne video vil vi vise dig, hvordan du laver en Spider -robot, som vi kan betjene ved hjælp af vores smartphone (Androi
Kontrol ledt over hele verden ved hjælp af internet ved hjælp af Arduino: 4 trin

Kontrol ledt over hele verden ved hjælp af internet ved hjælp af Arduino: Hej, jeg er Rithik. Vi kommer til at lave en internetstyret LED ved hjælp af din telefon. Vi kommer til at bruge software som Arduino IDE og Blynk.Det er enkelt, og hvis det lykkedes dig, kan du styre så mange elektroniske komponenter, du ønskerTing We Need: Hardware:
Sådan laver du en drone ved hjælp af Arduino UNO - Lav en quadcopter ved hjælp af mikrokontroller: 8 trin (med billeder)

Sådan laver du en drone ved hjælp af Arduino UNO | Lav en Quadcopter ved hjælp af mikrokontroller: Introduktion Besøg min Youtube -kanal En Drone er en meget dyr gadget (produkt) at købe. I dette indlæg vil jeg diskutere, hvordan jeg gør det billigt ?? Og hvordan kan du lave din egen sådan til en billig pris … Nå i Indien alle materialer (motorer, ESC'er
RF 433MHZ radiostyring ved hjælp af HT12D HT12E - Lav en RF -fjernbetjening ved hjælp af HT12E & HT12D med 433mhz: 5 trin

RF 433MHZ radiostyring ved hjælp af HT12D HT12E | Oprettelse af en RF -fjernbetjening ved hjælp af HT12E & HT12D med 433mhz: I denne instruktør vil jeg vise dig, hvordan du laver en RADIO -fjernbetjening ved hjælp af 433mhz sendermodtagermodul med HT12E -kode & HT12D -dekoder IC.I denne instruktive kan du sende og modtage data ved hjælp af meget meget billige KOMPONENTER SOM: HT
Trådløs fjernbetjening ved hjælp af 2,4 GHz NRF24L01 -modul med Arduino - Nrf24l01 4 -kanals / 6 -kanals sender modtager til Quadcopter - Rc Helikopter - Rc -fly ved hjælp af Arduino: 5 trin (med billeder)
Trådløs fjernbetjening ved hjælp af 2,4 GHz NRF24L01 -modul med Arduino | Nrf24l01 4 -kanals / 6 -kanals sender modtager til Quadcopter | Rc Helikopter | Rc -fly ved hjælp af Arduino: At betjene en Rc -bil | Quadcopter | Drone | RC -fly | RC -båd, vi har altid brug for en modtager og sender, antag at vi til RC QUADCOPTER har brug for en 6 -kanals sender og modtager, og den type TX og RX er for dyr, så vi laver en på vores
