Indholdsfortegnelse:
- Trin 1: Forudsætninger
- Trin 2: Forudsætninger (fortsat)
- Trin 3: Forudsætninger (fortsat)
- Trin 4: Forudsætninger (fortsat)
- Trin 5: Ryd Matlab op for at forberede kørselskoden
- Trin 6: Vælg 10 normale øjenbilleder og 10 billeder med diabetesretinopati symptomer
- Trin 7: Vælg 10 normale øjenbilleder og 10 billeder med diabetesretinopati symptomer (fortsat)
- Trin 8: Opret 2 variabler (normal og diagnosticeret), og sæt dem hver til 0
- Trin 9: Opret en for Loop for automatisk at uploade normale billeder
- Trin 10: Opret en for Loop for automatisk at uploade normale billeder (fortsat)
- Trin 11: Beskær billedets kanter
- Trin 12: Opret et gråtonebillede
- Trin 13: Opret et kontrastbillede
- Trin 14: Forbedr kontrastbilledet
- Trin 15: Opret et gennemsnitsfilter
- Trin 16: Kombiner det gennemsnitlige filter med det kontrasterede billede
- Trin 17: Lav en ny middelmaske ved at trække pixel fra
- Trin 18: Opret et binært filtreret billede
- Trin 19: Fjern mindre klatter fundet i de filtrerede billeder
- Trin 20: Opret et diskstruktureringselement
- Trin 21: Udfør morfologiske lukkeoperationer
- Trin 22: Find objekterne med forbindelse til mindst 8
- Trin 23: Find det maksimale antal forbundne pixels
- Trin 24: Indstil maks. Pixelværdier til 0, og find pixel med> = 26 pixelforbindelser
- Trin 25: Fjern blodkar i billedet
- Trin 26: Figurvisning
- Trin 27: Fjern fartøjer og tæl blodklatter
- Trin 28: Diagnostiser nethindebilledet baseret på antallet af identificerede blodpropper
- Trin 29: Hvis der er mere end 5 klatter …
- Trin 30: Gentag filtreringsprocessen for normale billeder med billedcifferværdier som 2 og 3
- Trin 31: Gentag hele processen for de diagnosticerede billeder
- Trin 32: Statistisk analyse
- Trin 33: Find tillidsinterval
Video: Automatiseret diagnose af diabetisk retinopati via MATLAB: 33 trin

2024 Forfatter: John Day | [email protected]. Sidst ændret: 2024-01-30 08:29

(Se kodeoversigt ovenfor)
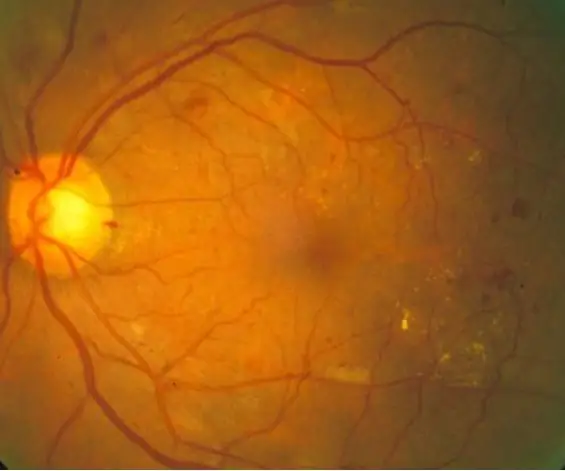
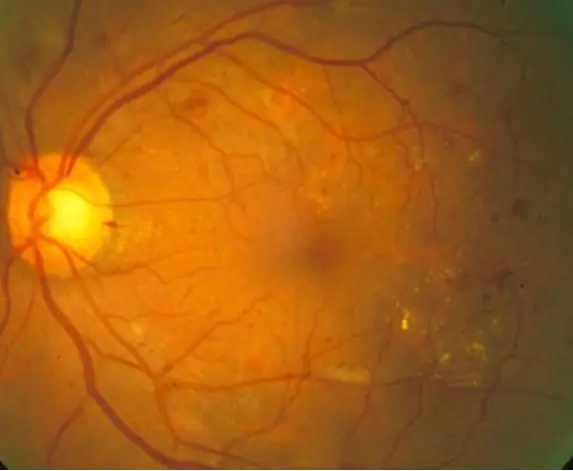
Diabetisk retinopati er en diabetesrelateret øjensygdom forårsaget af forhøjet blodsukker. De høje blodsukkerniveauer får blodkarrene i nethinderne til at svulme op, hvilket fører til forstørrede blodkar og endda karlækager, hvilket fører til mørke pletter i nethindebilleder. Med denne kode har vi til formål at bruge udseendet af lækager af blodkar som en indikator for diabetisk retinopati i baggrunden, selvom yderligere diagnoseteknikker ville være påkrævet i den virkelige verden. Målet med denne kode er at automatisere billedbehandling og diagnosticering af nethindebilleder for at identificere tegn på diabetisk retinopati vist gennem mørke pletter i nethindebillederne.
10 normale nethindebilleder og 10 diagnosticerede nethindebilleder blev behandlet gennem en kode, der først læser og filtrerer billederne og derefter kvantificerer de mørke pletter for at afgøre, om diabetiske retinopatisymptomer er til stede, baseret på en given tærskel. Resultaterne udskrives derefter på kommandovinduet for seertolkning.
Trin 1: Forudsætninger
1. Sørg for at have MATLAB -programmet downloadet til din computer.
2. Download txt -filen, der findes i linket. (Tryk på 'ctrl+s' for at gemme i det samme bibliotek som MATLAB -koden)
Trin 2: Forudsætninger (fortsat)
4. Åbn MATLAB, og skriv ‘uiimport’ i kommandovinduet.
5. Vælg filen officialdiagnoses.txt, og importer den til MATLAB som en cellematrix.
6. Sørg for at se 'officielle diagnoser' som en variabel i arbejdsområdet.
Trin 3: Forudsætninger (fortsat)

7. Download funktionen ModWald.m, som kan hentes fra ovenstående kode eller downloade den fra Canvas.
(Kode givet af professor King og professor Choi)
Trin 4: Forudsætninger (fortsat)

8. Download de 400 råbilleder fra datasektionen i STARE -projektet.
Trin 5: Ryd Matlab op for at forberede kørselskoden

Føj til kode:
1. luk alle (lukker alle tidligere åbnede billeder)
2. clearvars - undtagen officielle diagnoser (Sletter alle variabler undtagen den officielle diagnoser txt -fil, der tidligere er importeret)
3. cclc (sletter kommandovinduet)
Trin 6: Vælg 10 normale øjenbilleder og 10 billeder med diabetesretinopati symptomer
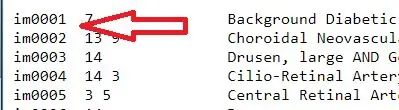
1. Tag diagnosetekstfilen og udtræk billednavnene. Disse navne er indeholdt i den første kolonne i tekstfilen, så for at udtrække dem skal du skrive 'officialdiagnoses (:, 1)'. Matrixen med billednavne blev tildelt en variabel, "alle_billede_numre"
2. Konverter variablen all_image_numbers fra et cellematrix til et matrix array ved hjælp af cell2mat -funktionen
Trin 7: Vælg 10 normale øjenbilleder og 10 billeder med diabetesretinopati symptomer (fortsat)
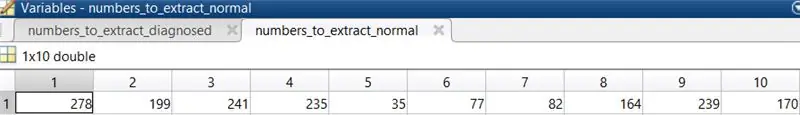
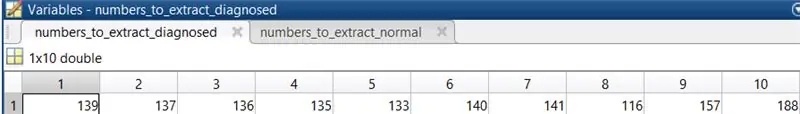
3. Vælg 10 normale øjenbilleder for at køre koden. De billeder, der blev valgt i denne sag, var 278, 199, 241, 235, 35, 77, 82, 164, 239, 170.
Placer disse tal i en matrix, og tildel dem til en variabel, der vil blive kaldt, når billederne indlæses.
4. Gentag trin 3 for nethindebilleder, der er blevet diagnosticeret med diabetisk retinopati. De billeder, der blev valgt i denne sag, var 139, 137, 136, 135, 133, 140, 141, 116, 157, 188.
Trin 8: Opret 2 variabler (normal og diagnosticeret), og sæt dem hver til 0

Opret disse variabler før for -løkken for at initialisere sløjfe -numrene.
Trin 9: Opret en for Loop for automatisk at uploade normale billeder

1. Opret en for loop
2. Indstil en tællevariabel (i, i dette tilfælde) til en matrix med værdier 1-10. Denne tællingsvariabel bruges til at kalde hvert billede individuelt
3. Tag i -elementet i billedmatrixen for at udtrække og konvertere billednavnet fra en streng til et tal ved hjælp af funktionen num2str.
Find antallet af cifre i billednavnet ved hjælp af numelfunktionen. Tildel denne værdi til en variabel, digits_normal. Dette tal skal være 1 for enkeltcifrede tal, 2 for tocifrede tal og 3 for trecifrede tal. Disse oplysninger bruges til automatisk at kalde billeder.
Trin 10: Opret en for Loop for automatisk at uploade normale billeder (fortsat)

3. Opret en if -sætning, der indeholder alle tre muligheder fra de foregående trin. Hvis billednavnet har 1 ciffer, vil billedet blive kaldt “im000”, hvis det har 2 cifre, vil billedet blive kaldt som “im00”, og hvis det har 3, vil billedet blive kaldt som “im0”.
4. Under hver if -sætning skal du tildele en variabel til at indlæse “im” under den tilsvarende if -sætning med det passende antal nuller (som beskrevet ovenfor) efterfulgt af i.
Trin 11: Beskær billedets kanter
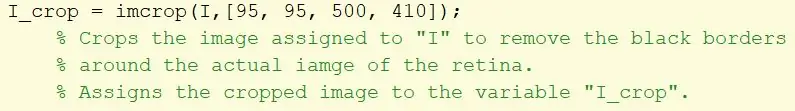
Tag det originale billede og anvend et imcrop -filter for at fjerne sorte kanter og tildele en variabel I_crop. Beskæringsrektanglet er angivet ved hjælp af en matrix [95, 95, 500, 410].
Trin 12: Opret et gråtonebillede

Tag det beskårne billede, og anvend rbg2gray -filteret for at ændre billedet til gråtoner. Tildel dette billede til variabel I2.
Trin 13: Opret et kontrastbillede
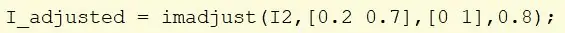
Tag billedet I2, og brug imadjust til at skalere intensitetsværdier.
Tag værdier, der falder inden for området [0,2, 0,7], og omskaler dem til en [0, 1]. Gamma er indstillet til 0,8 for at gøre billedet lysere. Tildel det nye billede til I_adjusted.
Trin 14: Forbedr kontrastbilledet
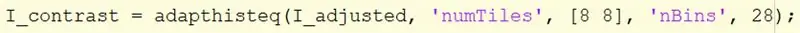
Tag det I_justerede billede, og brug adapthisteq -funktionen til at forbedre kontrasten.
Adapthisteq -syntaksen kræver billednavnet, I_adjusted, 'numTiles', størrelsen på numTiles, 'nBins' og antallet af bakker. Størrelsen på numTiles er indstillet til [8 8], billedet opdeles i 8x8 fliser, og antallet af skraldespande er sat til 28. Tildel billedet til I_constrast.
Trin 15: Opret et gennemsnitsfilter

Opret en variabel med navnet 'meanfilt' ved hjælp af fspecial -funktionen. Indtast 'gennemsnitsfunktion' for at oprette gennemsnitsfilteret og indsæt [90 90] for vinduesstørrelsen.
Trin 16: Kombiner det gennemsnitlige filter med det kontrasterede billede
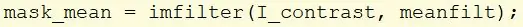
Opret en ny variabel ved navn mask_mean, og brug imfilter -funktionen til at tage I_contrast -billedet og anvende det gennemsnitlige filter, der tidligere blev oprettet.
Trin 17: Lav en ny middelmaske ved at trække pixel fra

Opret en variabel ved navn mask_mean2, og brug funktionen imsubtract til at trække værdien af hver pixel i I_contrast fra den tilsvarende pixel i mask_mean.
Trin 18: Opret et binært filtreret billede

Gør gråtonebilleder til sort / hvid ved hjælp af imbinarize. Input mask_mean2, 'adaptiv', 'Forgrundspolaritet', 'mørk', 'Følsomhed', 0.6. Tildel dette nye billede til mask_binarize.
Trin 19: Fjern mindre klatter fundet i de filtrerede billeder
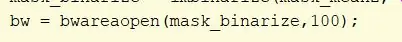
Fjern objekter med tilslutning mindre end 100 pixels ved hjælp af bwareaopen -funktionen på mask_binarize og indstil tærskelværdien til 100. Tildel variablen som bw.
Trin 20: Opret et diskstruktureringselement

Opret et diskstruktureringselement (med en radius på 2) ved hjælp af strel -funktionen. Tildel det til se.
Trin 21: Udfør morfologiske lukkeoperationer

Tag bw og anvend den tætte funktion på strukturelementet for at udføre en morfologisk tæt operation på objektet.
Trin 22: Find objekterne med forbindelse til mindst 8
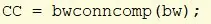
Tag bw og brug bwconncomp til at finde objekterne med tilslutning på mindst 8 i billedet. Tildel nummeroutput til cc_1.
Trin 23: Find det maksimale antal forbundne pixels
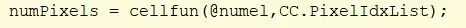
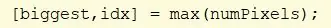
Brug cellfun -funktionen til at udføre funktionen "numel" på hver celle i CC. Dette finder antallet af elementer i PixelIdxList -cellen. Tildel værdi til "numPixels".
Find de maksimale værdier i numPIxels. Tildel det største maksimum til "største" og indekset for den maksimale værdi til "idx".
Trin 24: Indstil maks. Pixelværdier til 0, og find pixel med> = 26 pixelforbindelser
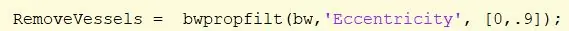
= 26 Pixel -forbindelse "src =" https://content.instructables.com/ORIG/FXY/DTW3/JEOIIEL4/FXYDTW3JEOIIEL4-p.webp

= 26 Pixel -forbindelse "src =" https://content.instructables.com/ORIG/FXO/GBX1/JEOIIELB/FXOGBX1JEOIIELB-p.webp

= 26 Pixel -forbindelse "src =" {{file.large_url | add: 'auto = webp & frame = 1 & height = 300' %} ">

= 26 Pixel -forbindelse "src =" {{file.large_url | add: 'auto = webp & frame = 1 & height = 300' %} ">
Indstil pixels med de største værdier i billedet “bw” til 0, hvilket gør pixelerne sorte.
Find objekterne med en forbindelse på mindst 26 pixels i billedet ved hjælp af bwconncomp. Tildel til variabel cc_1.
Trin 25: Fjern blodkar i billedet
Fjern de blodkar, der stadig er til stede på billedet, ved hjælp af bwpropfilt -funktionen med et område på [0, 0,9].
[0,9, 1] er udelukket, fordi værdier tæt på 1 angiver en linje. Tildel til “Fjern fartøjer”.
Trin 26: Figurvisning
Vis hvert filtreret billede i en underplot. Imshow. med 'border' og 'tight' input, viser hvert billede i en underplotstruktur. Tilføj en titel til hvert billede for at skelne mellem hvilket filter der blev brugt.
Trin 27: Fjern fartøjer og tæl blodklatter
1. Tag "RemoveVessels", og anvend funktionen "Centroid" i regionprops for at identificere centroiderne af objekterne i billedet. Disse objekter skal svare til blodpropper i billedet.
2. Tæl antallet af identificerede blodpropper ved at tage længden af centroidmatrixen.
Trin 28: Diagnostiser nethindebilledet baseret på antallet af identificerede blodpropper
Brug if -udsagn til at diagnosticere billede baseret på antallet af identificerede blodpropper.
Hvis antallet af identificerede centroider var mindre end eller lig med 5, blev billedet identificeret som normalt.
Hvis antallet af centroider var større end 5, blev billedet diagnosticeret med diabetisk retinopati.
Resultatet udskrives på kommandovinduet ved hjælp af fprintf.
Trin 29: Hvis der er mere end 5 klatter …
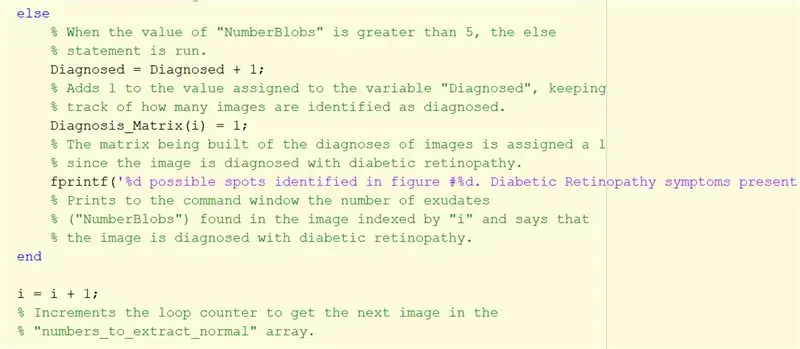
Gentag instruktionerne ovenfor for diagnosticerede billeder som en anden erklæring. Denne del kører, hvis antallet af klatter er større end 5.
Afslut if -sætningen.
Trin 30: Gentag filtreringsprocessen for normale billeder med billedcifferværdier som 2 og 3
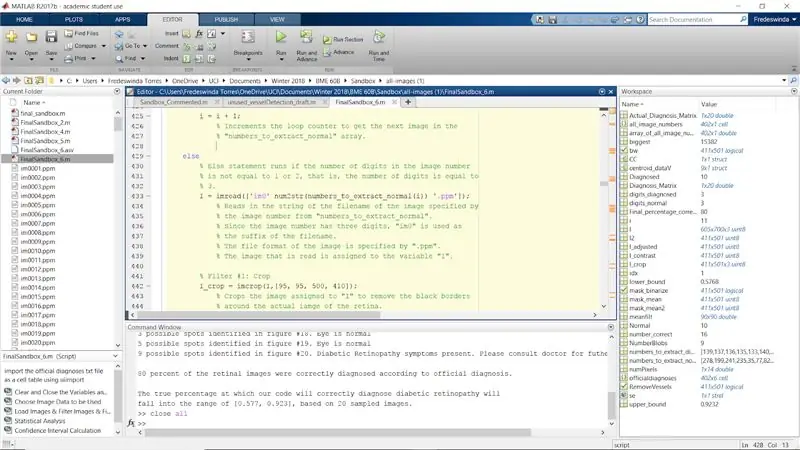
Gentag processen for resten af originalen, hvis udsagn, når numel (antallet af cifre i billednummeret) er lig med 2 og 3. Dette fuldender for -løkken for de normale billeder.
Afslut for -loop.
Trin 31: Gentag hele processen for de diagnosticerede billeder
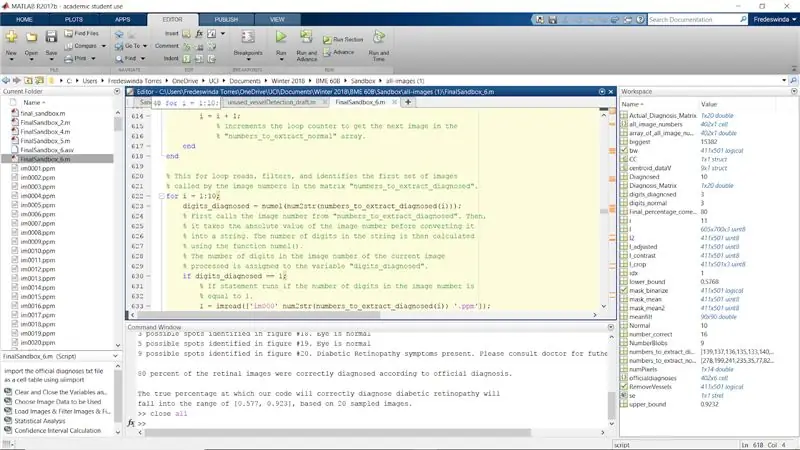
Gentag hele processen ved hjælp af de diagnosticerede billeder, der er angivet i matrixen "numbers_to_extract_diagnosed".
Sørg for at gå igennem hver figur (i) og ændre den til figur (i+10), så de diagnosticerede tal vil dukke op som billeder 11 til 20.
Trin 32: Statistisk analyse
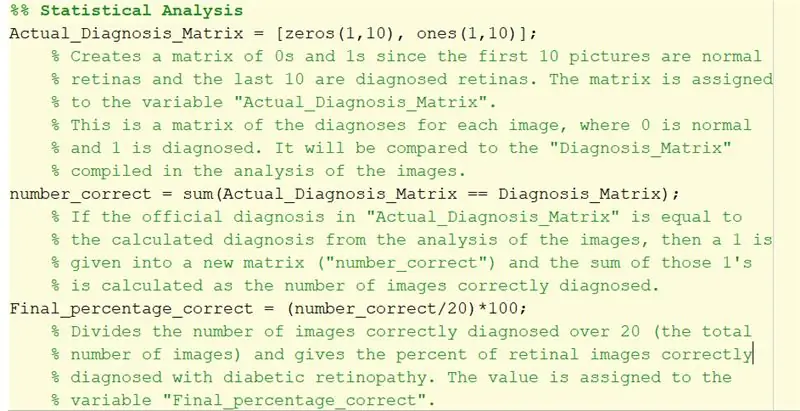
1. 'Actual_Diagnosis_Matrix' bruges til at sammenligne resultaterne med den officielle diagnose, der findes i txt -filen. De første 10 nuller angiver, at de første 10 billeder skal være normale. De sidste 10 viser, at de sidste 10 billeder skal klassificeres som diabetisk retinopati.
2. Det dobbelte lighedstegn, der bruges til at oprette 'number_correct', skaber et logisk array ved at sammenligne værdien af de tilsvarende elementer i 'Actual_Diagnosis_Matrix' med 'Diagnosis_Matrix', der er oprettet fra for -loop.
For hvert element, der matcher diagnosen, tilføjes 1, hvilket betyder, at koden korrekt diagnosticerede dette billede. Hvis det er forkert, tilføjer det et 0 til matricen.
Derefter tager summen af det sammen alle dem. Med andre ord finder den summen af korrekt diagnosticerede billeder.
3. 'Final_percentage_correct' er den beregnede procentdel af, hvor nøjagtig koden diagnosticerede diabetisk retinopati. Antallet af korrekt diagnosticerede billeder divideres med 20 (det samlede antal billeder) og ganges med 100 for at finde procentdelen af vellykkede diagnoser.
Trin 33: Find tillidsinterval
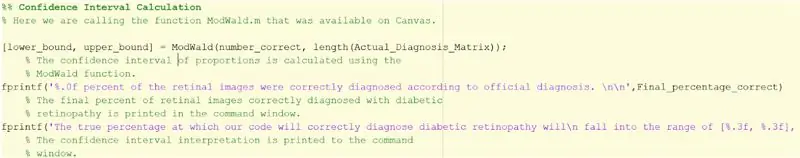
1. Sørg for at have ModWald.m downloadet for at kalde det som en funktion. Uden funktionen skulle du selv beregne konfidensintervallet ved hjælp af den modificerede Wald -metode.
2. ModWald -funktionen har 2 input, hvor den første er antallet af korrekt identificerede billeder, og den anden er den samlede mængde billeder.
3. ModWald -funktionen udsender de nedre og øvre grænser for konfidensintervallet for proportioner for nøjagtigheden af de samplede data. Med andre ord vil det give dig et interval af procenter, hvor den sande procentdel af nøjagtigheden af koden vil ligge.
4. Brug fprintf herunder til at udsende statistik og konfidensinterval til kommandovinduet.
> fprintf ('%. 0f procent af nethindebillederne blev korrekt diagnosticeret i henhold til officiel diagnose. / n / n', Final_percentage_correct)
> fprintf ('Den sande procentdel, ved hvilken vores kode korrekt diagnosticerer diabetisk retinopati, falder / n i området [%.3f, %.3f], baseret på 20 samplede billeder / n', lavere_bound, upper_bound)
Anbefalede:
Automatiseret EKG-BME 305 Slutprojekt Ekstra kredit: 7 trin

Automatiseret EKG-BME 305 Slutprojekt Ekstra kredit: Et elektrokardiogram (EKG eller EKG) bruges til at måle de elektriske signaler, der produceres af et bankende hjerte, og det spiller en stor rolle i diagnosen og prognosen for hjerte-kar-sygdomme. Nogle af de oplysninger, der er opnået fra et EKG, omfatter rytmen
Automatiseret EKG -kredsløbsmodel: 4 trin
Automatiseret EKG -kredsløbsmodel: Målet med dette projekt er at skabe en kredsløbsmodel med flere komponenter, der tilstrækkeligt kan forstærke og filtrere et indkommende EKG -signal. Tre komponenter vil blive modelleret individuelt: en instrumenteringsforstærker, et aktivt hakfilter og en
Automatiseret Pet-Food Bowl Project: 13 trin

Automated Pet-Food Bowl Project: Denne instruktive vil skildre og forklare, hvordan man bygger en automatiseret, programmerbar dyrefoder med vedhæftede madskåle. Jeg har vedhæftet en video her, der viser, hvordan produkterne fungerer, og hvordan det ser ud
Parkeringsassistent Nem reparation / diagnose: 4 trin

Parkeringsassistent Let at reparere / diagnosticere: Ok, lad os komme i gang, jeg har en 2010 Chevrolet Avalanche, og den har 4 parkeringsassistentsensorer i den bageste kofanger. Denne uoverkommelige kan bruges på og køretøj efter min bedste viden, vejret du har foran eller Rea eller begge dele. Så jeg gik til min favorit
Kontrolenheder via internettet via browser. (IoT): 6 trin

Kontrolenheder via internettet via browser. (IoT): I denne Instructable viser jeg dig, hvordan du kan styre enheder som lysdioder, relæer, motorer osv. Over internettet via webbrowser. Og du kan få adgang til kontrolelementerne sikkert over enhver enhed. Den webplatform, jeg brugte her, er RemoteMe.org -besøg
