Indholdsfortegnelse:
- Trin 1: Brug af Brain Box
- Trin 2: Intro: Analyse af tværsnit
- Trin 3: Opsætning af en funktion: LevelCurveTracings.m
- Trin 4: Udvikling af den bundne algoritme: Find grænser
- Trin 5: Udvikling af den bundne algoritme: Generering af ydre bundne array
- Trin 6: Udvikling af den bundne algoritme: Arbejde med centerpunkt
- Trin 7: Udvikling af den bundne algoritme: Når en Centroid ikke er centreret
- Trin 8: Udvikling af den bundne algoritme: Interferens af huller
- Trin 9: Udvikling af den bundne algoritme: Lokalisering af huller, afslutning af hjernegrænser og hulgrænser
- Trin 10: Logningsdata: Funktion PatientFiles.m
- Trin 11: Logning af data i filer
- Trin 12: Logningsdata: Visning af et plot af hjernevolumen over tid
- Trin 13: Lukning af huller i undergrunde: Subplotclose.m
- Trin 14: Hovedkoden: Sletning af alle og forespørgsel efter input
- Trin 15: Hovedkoden: Batchbehandling af billederne
- Trin 16: Hovedkoden: Polstring
- Trin 17: Hovedkoden: Bestemmelse af grænser
- Trin 18: Hovedkoden: Bestemmelse af den korrekte Z -dimension
- Trin 19: Hovedkoden: Bestemmelse af X- og Y -koordinater
- Trin 20: Hovedkoden: Plotte en tredimensionel struktur, finde volumen og logdata
Video: Brain Box: Tracking Neural Volume Over Time: 20 trin

2024 Forfatter: John Day | [email protected]. Sidst ændret: 2024-01-31 10:18
Fremskridtet til grænsen for længere menneskeliv har frembragt stigningen af sygdomme, som civilisationer ikke har set før vores. Blandt disse påvirkede Alzheimers cirka 5,3 millioner levende ældre amerikanere i 2017 eller cirka 1 ud af 10 ældre amerikanere (https://www.alz.org/facts/) og utallige andre med demens. For at hjælpe i kampen med at forstå, hvad der rammer vores ældste, vil denne kode udstyre de fremtidige forskere og ivrige nysgerrige med evnen til at spore hjernens volumen over tid.
Trin 1: Brug af Brain Box
For at bruge hjerneboks behøver man kun følgende:
- MR -scanninger af en hjerne og navn og format på sådanne filer (skal alle have nogenlunde de samme dimensioner)
- Længde på en scanning
- Afstand mellem hvert lag (MR -scanning)
- Patientnavn (Medtag ikke mellemrum, når du indtaster, og skriv venligst store for- og efternavne, f.eks.: Fornavn Efternavn)
Og ud fra dette har man evnen til at holde styr på individers tendenser i hjernens volumen over tid. Således kan tal for Alzheimers tendenser spores af denne software. Længden, vi brugte i forsøget, var 180 mm for længden af en scanning og 5 mm for afstanden mellem MR -scanninger, baseret på gennemsnitlige tal.
Imidlertid behøver anvendelsen af hjerneboks ikke at være begrænset til denne ene opgave. Hvis tværsnittene af et givet fast stof er fotografier, ligesom en tumor i sig selv, kan tendenserne i ændringer i volumen for disse også spores i softwaren.
Trin 2: Intro: Analyse af tværsnit
I tredimensionelle strukturer vil de todimensionelle planer, som sådanne består af, blive kaldt tværsnit. Forestil dig, at en stak papir udgør et rektangulært prisme, så ville hvert stykke papir være et tværsnit af papiret. Når vi forestiller os hjernen, anvender vi den samme tankegang. MR (magnetisk resonansbilleddannelse) (se information om MR) fanger hjernens tværsnit, og fra at bruge de grænser, der er defineret i hvert "lag" i hjernen, kan vi konstruere en struktur til at modellere og finde hjernens volumen. Vi skal dog først opbygge en funktion til at give information om sådanne grænser.
Trin 3: Opsætning af en funktion: LevelCurveTracings.m
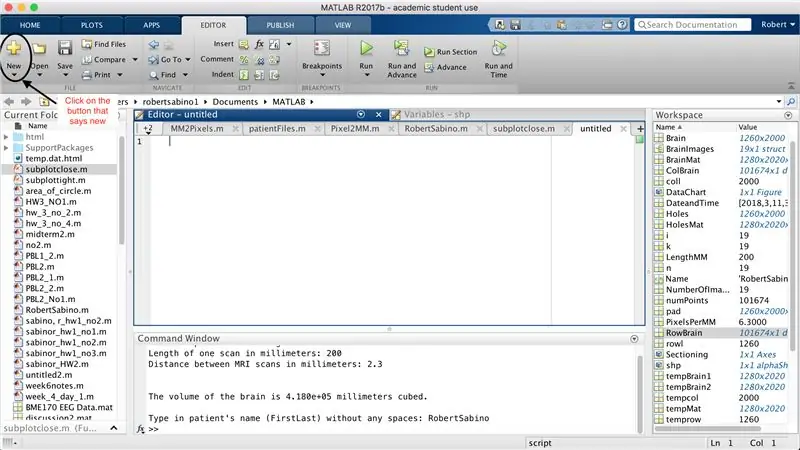
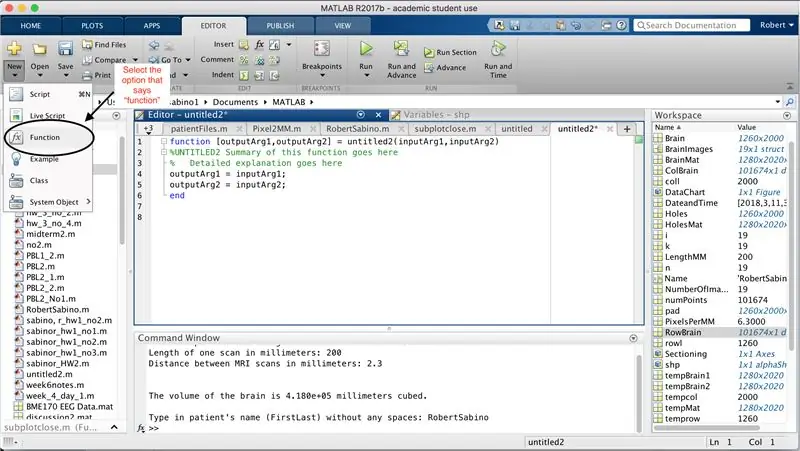
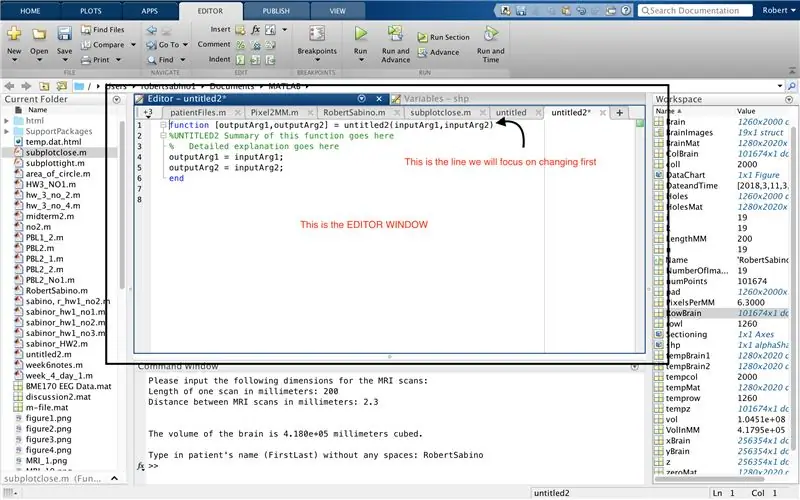

Sørg først for, at din computer har MATLAB_R2017b downloadet (download her), og åbn MATLAB. I MATLAB -grænsefladen skal du klikke på knappen i øverste venstre hjørne af vinduet, der siger "Ny" med et fedt gult plustegn, og vælg indstillingen "funktion" for at åbne et redigeringsvindue et mellemrum, der ligner det i tredje billede. Vi vil fokusere på at ændre den første linje for at konfigurere funktionen. Hvor der står "outputArg1", skal du erstatte det med "brain", "outputArg2" for at sige "huller", "untitled2" til "exp2" og "inputArg1" til "image" og slette "inputArg2". Du har nu en funktion, der skal kaldes ved hjælp af "exp2", idet du tager et argument "billede" og udsender grænserne for "hjerne" og "huller". Den første linje i funktionen skal ligne den linje, der er afbildet på det fjerde billede. Slet hele koden under denne indledende linje.
Trin 4: Udvikling af den bundne algoritme: Find grænser

Indtast koden som følger under linjen. Denne sektion af funktionen udfører følgende linje-for-linje.
- Indlæs billedet "billede" i variablen "mri".
- Gør "mri" til et billede, der består af værdier i en række tal til enere og nuller (alias binærisering) baseret på en fastsat tærskelværdi. Hvis værdien i en pixel er lig med eller større end 0,1, sættes den til en, hvis ikke, er værdien ved denne pixel sat til nul.
- De følgende fire linjer omdanner 10 kolonner og rækker i kanterne af MR -scanningslaget til nuller for at undgå at læse forkerte værdier som grænser (som man lærte ved at eksperimentere med koden).
- I den sidste linje sporer bwboundaries grænserne for det binære billede "mri" og sætter det lig med "b", en matrix med de elementer, hvis indekser svarer til grænserne, der er sat til et.
Trin 5: Udvikling af den bundne algoritme: Generering af ydre bundne array
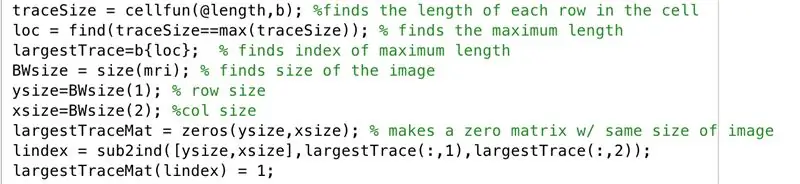
Følg med i editorvinduet med følgende kode på billedet. Dette afsnit af koden gør følgende linje-for-linje.
- Find længden af hver af rækkerne i det binære billede "b" (cellfun anvender funktionslængden til hver række).
- Indstil "loc" for at gemme de maksimale længder.
- Find indekset for den maksimale længde, indstillet til at gemme i "LargestTrace".
- Find størrelsen på billedet "mri", som består af samme størrelse som "b", og indstil til "BWsize".
- Find antallet af rækker i billedets array, indstillet til "ysize".
- Find antallet af kolonner i billedets array, indstillet til "xsize".
- Generer array "grootsteTraceMat", en "ysize" ved "xsize" matrix af nuller.
- Find det ækvivalente indeks fra de abonnerede værdier, der svarer til, hvor de størsteTrace x -værdier og y -værdier var, gem i vektoren "lindex".
- I matricen med nuller, "LargestTraceMat", skal du omsætte de elementer ved indekserne, der svarer til indeksværdier, der er gemt som elementer i "lindex", til dem.
Således har det logiske array "LargestTraceMat" det største afgrænsede område af det givne hjernescannetværsnit aftegnet som dem med baggrund af nuller
Trin 6: Udvikling af den bundne algoritme: Arbejde med centerpunkt

Dernæst skal vi teste, om tværsnittet består af mere end en region (den største). Ved at teste justeringen af den største regions centroid kan vi se, om der er en sammenhængende region, som ville give en mere centreret centroid eller muligheden for flere regioner.
- Brug "regionProps" til at finde oplysninger om de tilstedeværende centroider, der er lig med strukturmatrixen "tempStruct"
- Form array "centroids" med data fra feltet "centroid" sammenkædet lodret
- Tag den anden kolonneværdier for "centroids" (de vandrette dimensionskoordinater)
- Kør et filter for at kontrollere centroidens tilpasning til det vandrette center
Trin 7: Udvikling af den bundne algoritme: Når en Centroid ikke er centreret
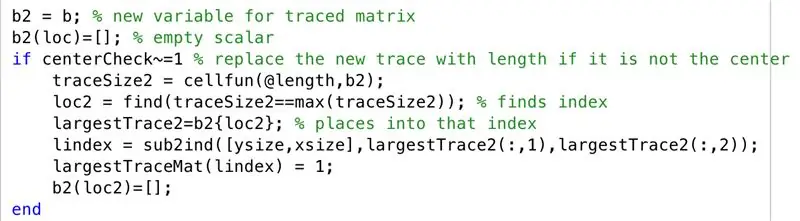
I scenariet, hvor centroiden for det største sporområde ikke er centreret, gennemgår vi følgende trin. Som vi havde observeret i MR -scanningerne, var tendensen at få hjernehalvdele afbildet i tværsnittet, når de ikke var sammenhængende, så vi fortsætter nu med at plotte det næststørste spor sammen med det største spor i "grutsteTraceMat"
- Indstil den sporede matrix til en ny variabel "b2"
- Initialiser tom matrix "b2", med et sæt indekseret af "loc"
- Opret en betinget, for når en centroid ikke er centreret (dvs. et lag med flere regioner)
- Indstil en ny sporstørrelse, der skal findes for hver række (traceSize2)
- Indstil "loc2" for at finde de indekser, hvor grænser er til stede
- Lad celler specificeret med "loc2" i "b2" være lig med "grutsteTrace2"
- Konverter abonnementer til indekser, indstillet til "lindex"
- Skift elementer svarende til "lindex" i "LargestTraceMat" til 1
- Initialiser tom matrix "b2", med et sæt indekseret af "loc2"
Trin 8: Udvikling af den bundne algoritme: Interferens af huller

Ved håndtering af huller holdt de værdier, der er gemt i "b2" styr på andre strukturer end det største spor, og afbildning af disse på en fyldt form af "grutsteTraceMat" afslører, hvor der er huller i hjerneområderne.
- Opret array "filledMat", som er en udfyldt i form af "grutsteTraceMat"
- Opret array "interferenceMat", en "ysize" ved "xsize" array af nuller
- Opret array "interferenceloc" for at gemme værdierne fra "b2", sammenkædet lodret
- Opret array "lindex" for at gemme de indeks, der svarer til "interferenceloc"
- For indeks i "interferenceMat", der svarer til "lindex", sættes værdien til 1, hvilket gør en anden afgrænset region
Trin 9: Udvikling af den bundne algoritme: Lokalisering af huller, afslutning af hjernegrænser og hulgrænser

- Indstil array "tempMat" lig med "interferenceMat" plus "filledMat", og tilføj således hver værdi i matrixen til hinanden
- Indstil array "holesLoc" lig med indekserne, hvor "interferenceMat" og "filledMat" begge var lig med en
- Opsæt "holesMat" som en nulmatrix med dimensioner "ysize" x "xsize"
- Indstil indekser i "holesMat", der er lig med "holesLoc" som dem
- Indstil "hjerne" til "grutsteTraceMat"
- Indstil "huller" til "holesMat"
Med fundet, hvor værdierne af de tilføjede matricer var lig med 2, blev hullets placeringer let sikret og afbildet på en tom matrix.
Trin 10: Logningsdata: Funktion PatientFiles.m
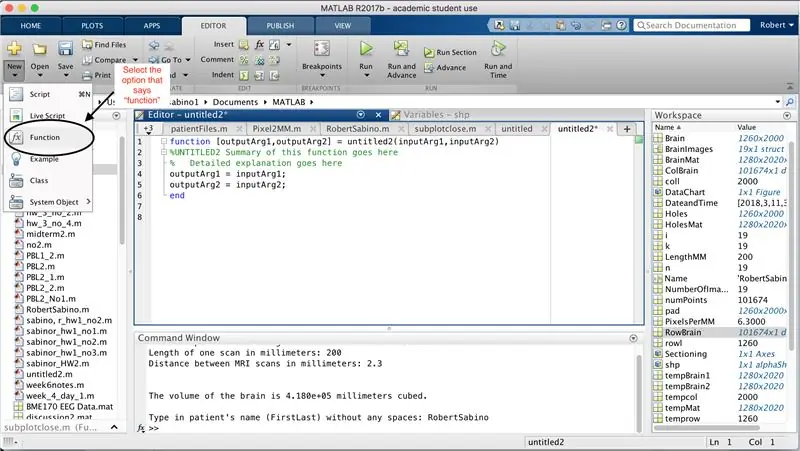
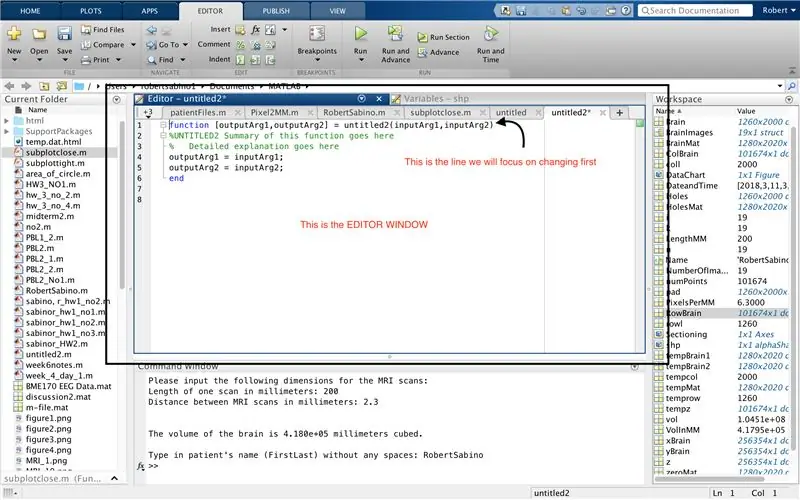

Ligesom opsætningen af den sidste funktion, skal du klikke på knappen i øverste venstre hjørne af vinduet, der siger "Ny" med et fedt gult plustegn, og vælg indstillingen "funktion" for at åbne et mellemrum i editorvinduet ligner det på det tredje billede. I den første linje skal du slette outputmatricen og erstatte med blot "output", erstatte "untitled2" med "patientFiles", slette alle inputargumenterne og i stedet følge den formatering, der er angivet i det fjerde billede af kodelinjen. Den første linje i denne funktion skal matche billedets formatering.
Trin 11: Logning af data i filer
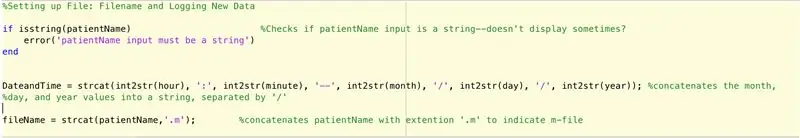

For at oprette en fil til at logge de data, der findes af hovedfunktionen (endnu ikke beskrevet), skal vi følge disse trin (som foreskrevet af koden linje for linje).
- Kontroller, om input til patientnavn er en streng.
- Hvis det ikke er en streng, skal du vise, at patientName -input skal være en streng.
- Afslut if -sætningen (forhindr fejl).
- Opret en streng sætning "DateandTime", der vil give følgende format: time: minutter-måned/dag/år.
- Indstil variabel filnavn til følgende: patientnavn.m.
Nu til det næste afsnit af funktionen: Findes der allerede en fil med dette navn?
1) Antag, at filen med dette navn allerede findes:
- Kør filen for at få værdierne fra fortiden i kø
- Tilføj dataene "DateandTime" for den aktuelle iteration som en ny celle i cellearrayet med x -værdier (indeksende+1)
- Tilføj den aktuelle "brainVolume" -værdi som en ny celle i cellearrayet med y -værdier (indeksende+1)
- Gem de aktuelle variabler, der er indlæst i filen.
2) Antag, at filen med dette navn ikke findes:
- Opret en ny fil med navnet gemt i variablen "patientnavn"
- Tilføj de aktuelle "DateandTime" -data som en celle i det tomme cellearray med x -værdier
- Tilføj de nuværende "brainVolume" -data som en celle i det tomme cellearray med y -værdier
- Gem de aktuelle variabler, der er indlæst i filen.
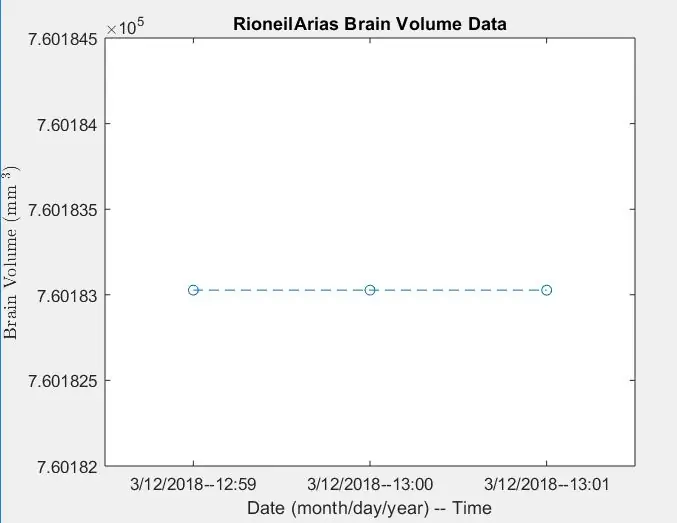
Trin 12: Logningsdata: Visning af et plot af hjernevolumen over tid

- Konverter x -værdier array (xVals) til et kategorisk array (xValsCategorical), for at tillade plotting
- Generer figurvindue 5
- Plot punkterne angivet med "xValsCategorical" og "yVals" (indeholdende hjernevolumen) ved hjælp af hule cirkler til at angive punkter og blive forbundet med stiplede linjer
- Titel plottet som: patientName Brain Volume Data
- Mærk x -aksen som vist på billedet
- Mærk y -aksen som vist på billedet
- Lad figur 5 svare til output
Fra dette vil funktionen patientnavn, der kaldes, give en fil med redigerede data, der holder styr på hjernens volumen over tid og et plot, der viser tendenser.
Trin 13: Lukning af huller i undergrunde: Subplotclose.m

Funktionen, tilpasset fra kode fra https://www.briandalessandro.com, fungerer for at lukke hullerne mellem hovedplodens underplotfigurer, når figurerne, der viser MR -billederne og hjernelagene, oprettes. Subplot -funktionen, der bruges inden for subplotclose.m, justerer placeringen af de givne subplots, så de passer godt til hinanden i aspektet af den længere dimension. For eksempel, hvis koden har til hensigt en 7 x 3 matrix, passer rækkerne tæt, da rækkedimensionen er længere. Hvis koden har til hensigt en 3 x 7 matrix, passer kolonnerne tæt, med huller i rækkerne, som vist i figurerne i vores hovedkode.
Trin 14: Hovedkoden: Sletning af alle og forespørgsel efter input

For at starte hovedkoden skal du klikke på den samme knap, der siger "Ny" i øverste venstre hjørne af vinduet, og vælge "Script" i stedet for "Funktion" fra de tidligere sektioner. Indtast koden som vist på billedet i editorvinduet. Kodelinjerne udfører følgende opgaver i rækkefølge:
- Luk alle åbne filer undtagen 0, 1 og 2.
- Luk alle figurvinduer.
- Ryd alle variabler i arbejdsområdet.
- Ryd kommandovinduet.
- Visning i kommandovinduet: Angiv følgende dimensioner for MR -scanninger:
- På en ny linje i kommandovinduet, spørg: Længde af en scanning i milimeter:. Svaret fra brugeren indstilles til variablen "lengthMM".
- Spørg på en ny linje: Afstand mellem MR -scanninger i milimeter:. Svaret fra brugeren indstilles til variablen "ZStacks".
Trin 15: Hovedkoden: Batchbehandling af billederne

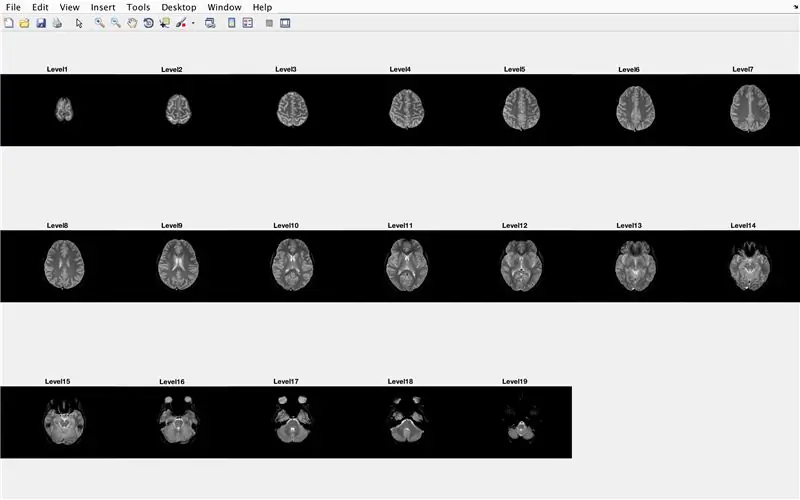
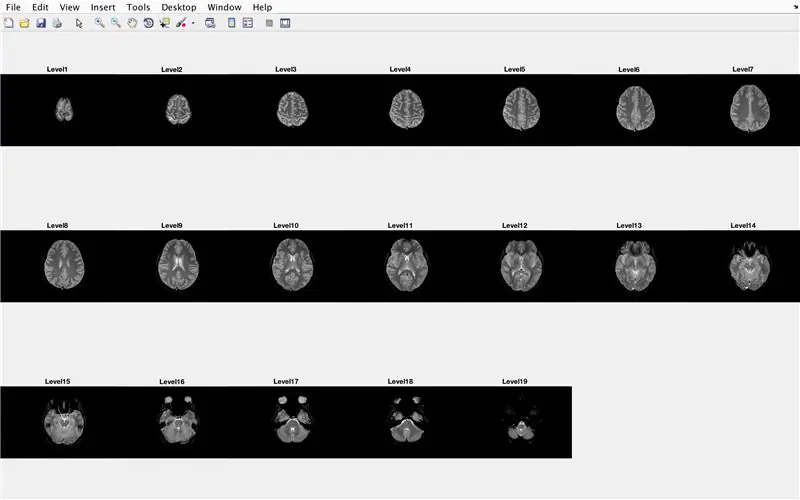
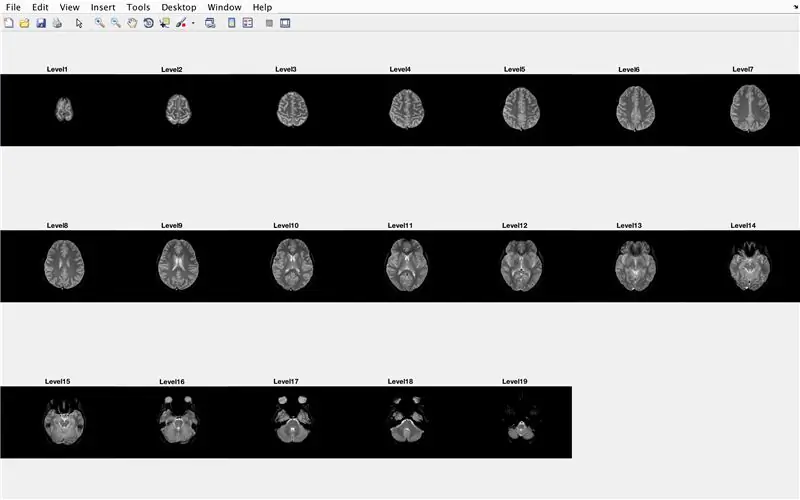
I dette afsnit vil koden indlæse billederne (bestående af MR -scanninger af hjernens tværsnit) og gemme navnene på hver billedfil i variablen "Base" og vise hver af MR -scanninger. Følg venligst med koden på billedet, som gør følgende:
- Opret struktur array "BrainImages", der indeholder oplysninger om alle filer i den aktuelle mappe, der passer til navneformatet for MRI _ (). Png
- Indstil variabel "NumberofImages" lig med antallet af elementer i strukturmatrixen "BrainImages"
- Åbn figurvindue 1
- Indstil en for -loop til at gennemgå antallet af billeder, der tælles i filen
- For hver sløjfe er "CurrentImage" det respektive navn på hver fil MRI_i-p.webp" />
- Generer en 3 x 7 delplot for at vise de 19 billeder, der skal indlæses af "imshow"
- Vis hvert billede som et andet element i underplotfigurvinduet
- Titel hvert delplot -element som niveau_, hvor tomt er iterationsnummeret for for -løkken.
- Afslut for -loop (undgå fejl)
Dette viser i figurvindue 1 alle MR -scanninger i rå form i en 3 x 7 -konfiguration uden huller i x -orienteringen.
Trin 16: Hovedkoden: Polstring

Med polstring undgår vi problemet med små uoverensstemmelser i billedstørrelserne, der kan give fejl ved dimensionens uoverensstemmelse, hvis et billede er lidt større end et andet.
- Åbn figurvindue 2
- Indlæs billedmatrixen fra MRI_1-p.webp" />
- Find størrelsen på billedets matrix og indstil til "OriginalXPixels" (for antal rækker) og "OriginalYPixels" (for antal kolonner)
- Opsæt matrixen "BrainMat" til at bestå af alle nuller med 20 flere rækker og 20 flere kolonner for hvert plan og 19 samlede tværsnit, en pr. Plan.
- Konfigurer "HolesMat" til at bestå af det samme tredimensionale array af nuller til senere at indtaste hulkoordinater
- Opret "zeroMat" for at være på størrelse med pad plus tyve rækker og tyve kolonner, et todimensionalt array af nuller.
Trin 17: Hovedkoden: Bestemmelse af grænser
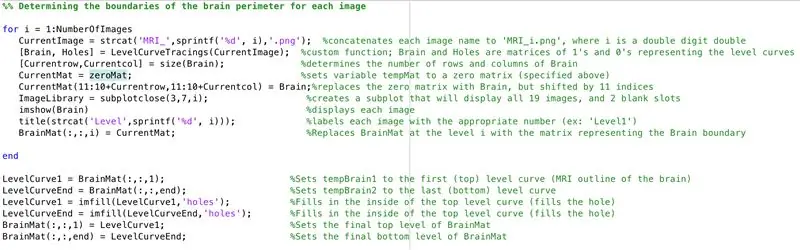
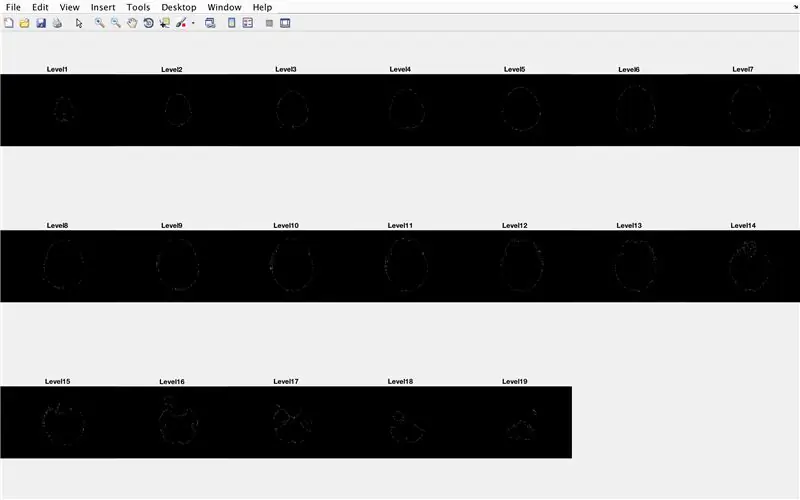
- Indstil en for loop til at gå igennem dataene fra hvert billede, der er indlæst tidligere
- På samme måde som batchbehandling tidligere, indlæser "CurrentImage" filer med "MRI_i.png", hvor i er iterationsnummeret
- Kør hvert billede gennem behandlingsfunktionen "LevelCurveTracings2.m", som du lavede tidligere
- Find størrelsen på output "Brain", sæt antallet af rækker til "Currentrow" og antallet af kolonner til "Currentcolumns"
- Indstil "CurrentMat" til en matrix af nuller med de dimensioner, der er angivet af "Currentrow" og "Currentcolumns"
- Centrer dataene fra "Brain" i "CurrentMat" med en margen på 10 rækker på alle sider
- Generer en underplot med dimensioner 3 x 7, for at vise billedernes grænser
- Titel på hvert af delplotelementerne i figurvinduet
- Generer tredimensionel matrix "BrainMat" bestående af hvert grænselag "CurrentMat"
- Afslut for -loop (for at undgå fejl)
Underafsnittet efterfylder hullerne i toppen og bunden af den foreslåede tredimensionelle form
- Indstil "LevelCurve1" lig med det første lag af "BrainMat" (bunden af fast stof)
- Indstil "LevelCurveEnd" lig med det sidste lag af "BrainMat" (øverst på solid)
- Overskriv "LevelCurve1" med et udfyldt lag
- Overskriv "LevelCurveEnd" med et udfyldt lag
- Indstil det udfyldte lag som det nederste lag af "BrainMat"
- Indstil det udfyldte lag som det øverste lag af "BrainMat"
Trin 18: Hovedkoden: Bestemmelse af den korrekte Z -dimension

De tre første linjer består i at oprette et tomt array "z" og udføre enkle konverteringsoperationer (divider pixel med længde) for at få en korrekt aflæsning af volumen i mm^3
- Opret en for -loop for at cykle gennem hvert lag
- Find antallet af dem i et givet lag
- Konverter z -koordinaterne for dem til værdier, der skaleres til det korrekte forhold, indstillet til "tempz", en kolonnevektor
- Tilføj z -værdi for niveaukurven til vektoren z
Med denne z koordinater justeres korrekt.
Trin 19: Hovedkoden: Bestemmelse af X- og Y -koordinater

Nu for at bestemme x- og y -positionerne for hvert af punkterne i grænserne.
- Initialiser "xBrain" som et tomt array
- Initialiser "yBrain" som et tomt array
- Indstil en for loop til at gå gennem hvert indlæste billede
- Kompilér en matrix med to kolonner for at gemme de plane koordinater for hvert punkt i bunden, repræsenteret af kolonnevektorerne "RowBrain" og "ColumnBrain"
- Tilføj "xBrain" med de aktuelt fundne "RowBrain" -koordinater
- Tilføj "yBrain" med de aktuelt fundne "ColumnBrain" -koordinater
- Afslut for -loop (for at undgå fejl)
Trin 20: Hovedkoden: Plotte en tredimensionel struktur, finde volumen og logdata
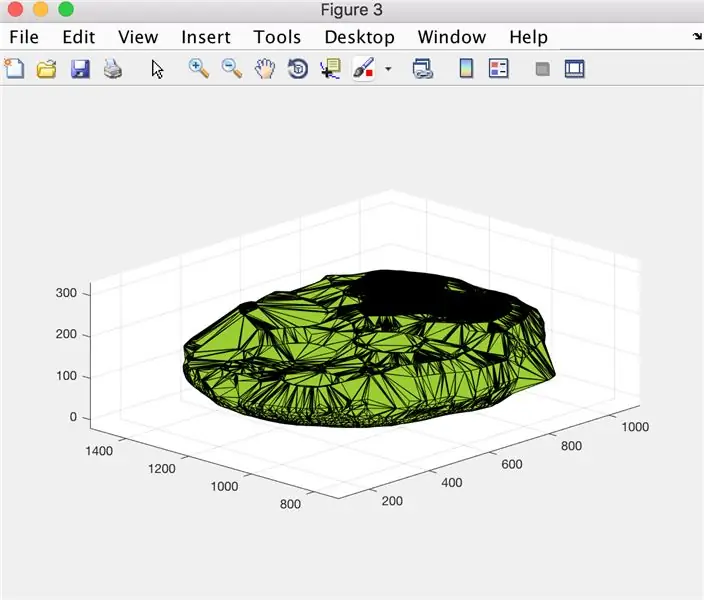

Ved hjælp af funktionen alphaShape vil vi oprette en tredimensionel struktur, hvorfra vi kan beregne hjernens volumen.
- Brug funktionen alphaShape, tilslut vektorerne "xBrain", "yBrain" og "z" for x-, y- og z -koordinaterne, og sæt lig med "BrainPolyhedron"
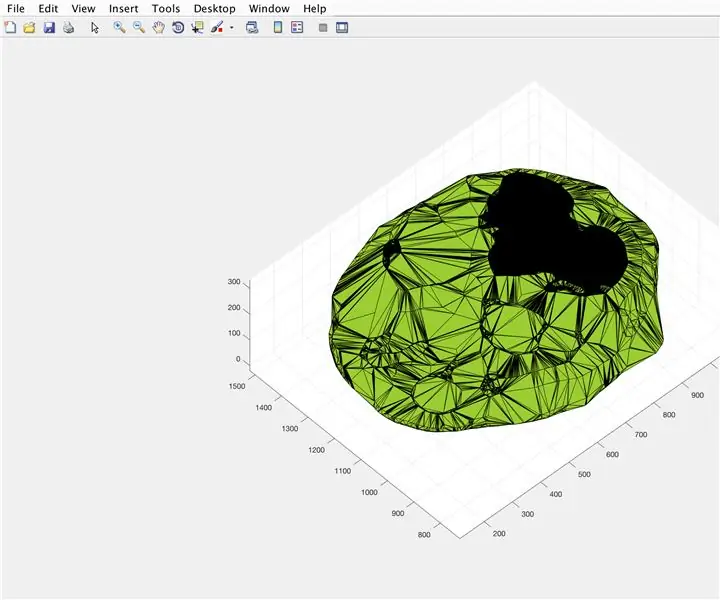
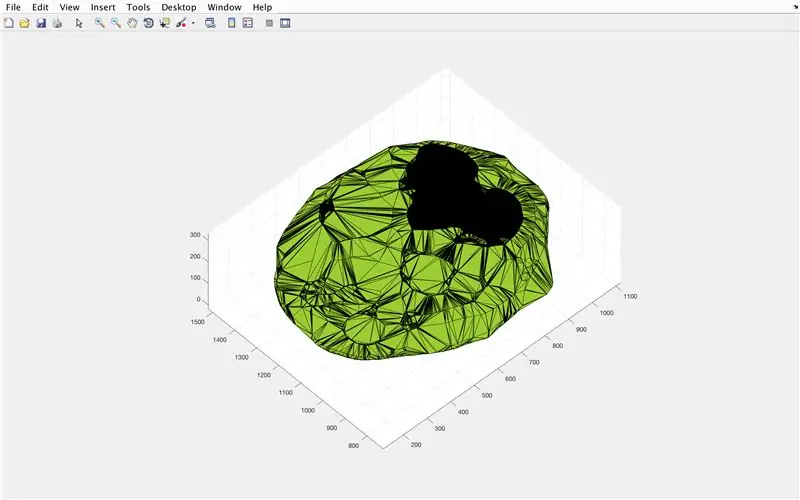
- Åbn figurvindue 3
- Plot den beregnede alfa -form "BrainPolyhedron", der vises i figurvinduet
- Beregn volumen af alfa -formen ved hjælp af en funktion "volumen", der fungerer for alfa -former
- Konverter lydstyrken til mm^3
- Udskriv mængden af det faste stof i kommandovinduet
- Bed om, at et patientnavn skal indstilles som input
- Få den aktuelle dato og klokkeslæt med uret, og indstil til "DateandTime"
- Opkaldsfunktion "patientFiles" for at logge og plotte de beregnede data
Herfra skal det andet og tredje billede vise de figurer, der dukker op, og det fjerde billede det, der skal vises i kommandovinduet.
Anbefalede:
DIY Smart Robot Tracking Car Kits Tracking Car Fotosensitive: 7 trin

DIY Smart Robot Tracking Car Kits Tracking Car Fotofølsom: Design af SINONING ROBOT Du kan købe fra tracking robot carTheoryLM393 chip sammenligne de to fotoresistor, når der er en fotoresistor LED på den ene side på HVID, stopper motorens side med det samme, den anden side af motoren drej op, så
Time Cube - Arduino Time Tracking Gadget: 6 trin

Time Cube - Arduino Time Tracking Gadget: Jeg vil gerne foreslå dig et enkelt, men virkelig nyttigt arduino -projekt til at spore tidsbegivenheder ved at vende en smart cube -gadget. Vend det til " Arbejde " > " Lær " > " Opgaver " > " Hvile " side, og det vil tælle
3 fantastiske BRAIN / MIND -kontrolprojekter Lights LedStrip LED med Arduino og Neurosky: 6 trin (med billeder)

3 fantastiske BRAIN / MIND -kontrolprojekter Lights LedStrip LED med Arduino og Neurosky: Har du nogensinde ønsket at tænde eller slukke lysene bare ved at tænke over det? Eller vil du vide, hvor stresset du er ved at se på farven på en RGB -led? Mens nu kan du ved at følge denne instruktion! For at få en fornemmelse af, hvad vi vil
Er det en hånd? (Raspberry Pi Camera + Neural Network) Del 1/2: 16 trin (med billeder)

Er det en hånd? (Raspberry Pi Camera + Neural Network) Del 1/2: For et par dage siden skadede jeg mit højre håndled i gymnastiksalen. Bagefter hver gang jeg brugte min computermus, forårsagede det mange smerter på grund af stejl håndledsvinkel. Det var da det ramte mig " ville det ikke være fantastisk, hvis vi kunne konvertere enhver overflade til en trackp
Arduino Neural Network Robot: 21 trin (med billeder)

Arduino Neural Network Robot: Denne instruerbare er baseret på en 3 -delt serie, jeg lavede til Make YouTube Channel, som viser dig præcis, hvordan du prototype, designer, samler og programmerer din egen Arduino neurale netværksrobot. Efter at have set hele serien, skal du have en bette
