Indholdsfortegnelse:
- Trin 1: Organisering af mammogramdata
- Trin 2: Billedbehandling
- Trin 3: Tærskelkode
- Trin 4: Find abnormiteter for hvert binært billede
- Trin 5: Planlægning af den diagnosticerede masseplacering og -størrelse til visuel sammenligning
- Trin 6: Implementering af den anden sammenligningsmetode
- Trin 7: Analyse af indsamlede data
- Trin 8: Lav din egen klassificering
- Trin 9: Forbedringer? Nogen tanker?
Video: Brug af varierende gråtonetærskler til at visualisere og identificere abnormiteter i mammogrambilleder: 9 trin

2024 Forfatter: John Day | [email protected]. Sidst ændret: 2024-01-30 08:28
Formålet med dette projekt var at identificere og bruge en parameter til at behandle gråtonemammogrammbilleder af forskellige baggrundsvævsklassifikationer: Fedt, fedtkirtel og tæt væv. Denne klassifikation bruges, når radiologer analyserer mammogrammer og skal overveje, om vævstæthed vil skjule eventuelle abnormiteter, såsom læsioner eller tumorer. Dette skyldes, at både normale fysiologiske strukturer, såsom kirtelvæv og fibrøst bindevæv. og unormale morfologier som forkalkninger og tumorer vil fremstå meget lyse på mammogrammet, mens mindre tæt fedtvæv vil se sort ud. Derfor var det passende at programmere en klassifikator, der kan manipulere pixelintensitetsniveauer for bedst at visualisere og identificere masser.
Trin 1: Organisering af mammogramdata
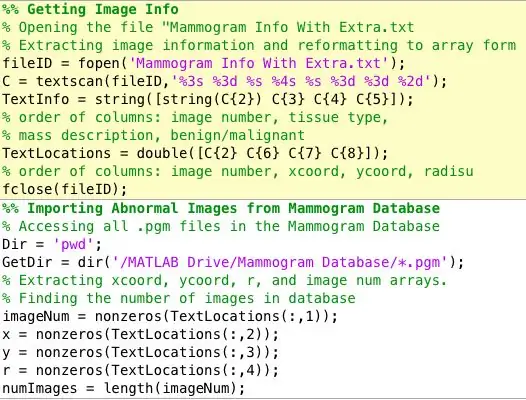
En af de første ting, jeg indså, at jeg skulle håndtere, var at organisere dataene på en meget klar, kortfattet og tilgængelig måde. Dette er de variabler, jeg hentede fra mini-MIAS-databasen over mammogrammer. Jeg lavede to arrays. En der indeholder 4 kolonner:
- Billednummer:
- x massekoordinat
- y massekoordinat
- Masseradius: (Dette definerede en omtrentlig størrelse for massen
Det andet array indeholdt klassificeringsinformation:
- Type baggrundsvæv: Fedtet (F), Fedtkirtlen (G), Tæt (D)
- Beskrivelse af masse: Veldefineret (CIRC), spikuleret (SPIC), dårligt defineret anden (MISC) Arkitektonisk forvrængning (ARCH), Asymmetri (ASYM), Normal (NORM)
- Diagnoser: Godartet (B), ondartet (M)
Da formålet med dette projekt var at bestemme den bedste tærskel for hver type baggrundsvæv, var ikke alle oplysninger nødvendige. Du kan dog udvide dit projekt til at omfatte teksturanalyse og teste din klassifikator mod de kendte massebeskrivelser.
Sidebemærkning: Databasen, hvorfra jeg fik de diagnosticerede mammogrambilleder, organiserede oplysningerne om hvert mammogram i en tekstfil adskilt fra billederne. Det var mildt sagt svært for mig at udtrække dataene fra en tekstfil og organisere i matrixformularer, men det følgende link var meget nyttigt til at finde ud af alt det. Alternativt kan du bare justere den kode, jeg har indsat ovenfor til dine formål.
Mammogramfilformat: mdb001 G CIRC B 535 425 197
mdb002 G CIRC B 522 280 69
TextScan Hjælp: https://www.mathworks.com/help/matlab/ref/textsca…Mammogram Database:
Trin 2: Billedbehandling
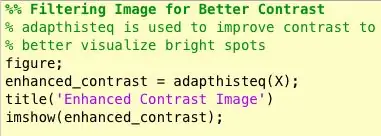
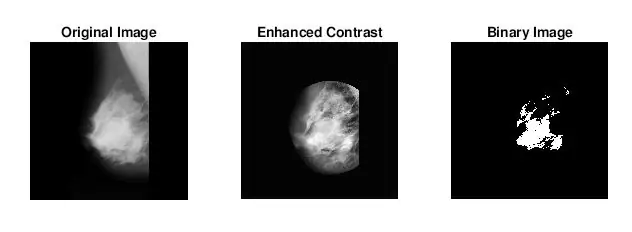
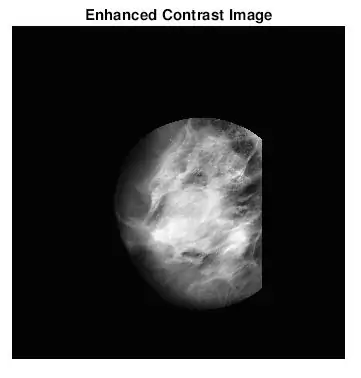
Den anden ting, der dukkede op, da jeg var ved at finde ud af at identificere masser, var, at jeg for mange unormale mammogrammer ikke visuelt kunne se, hvor abnormiteten var, eller hvor stor den var. Da jeg ikke er en erfaren radiolog, var det naturligvis forventet. Den mest ligetil måde at finde abnormiteter på (ifølge mine lange google -søgninger) var imidlertid at se på koncentrationer af lyse og mørke områder. Jeg brugte primært adapthisteq -funktionen til at forbedre billedkontrasten og derefter imbinarisere for at konvertere billedet til et binært billede for at eksperimentere med forskellige tærskelniveauer.
- adapthisteq: Denne funktion transformerer intensitetsværdier for gråtoner og rgb -billeder ved hjælp af kontrastbegrænset adaptiv histogramudligning. Med andre ord justerer det histogrammet for intensitetsværdier til en bestemt distributionstype. Matematik -linket til denne funktion er vedhæftet nedenfor for yderligere læsning.
- imbinarize: opretter et binært billede fra et gråtonebillede ved at tildele alle pixels over en bestemt intenisti til 1s og pixelerne under denne værdi a 0. Jeg brugte denne funktion til at teste den optimale tærskel for at reducere baggrundsvævsstøj.
Trin 3: Tærskelkode
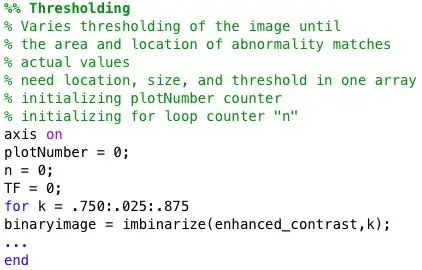
En for loop bruges til at binarisere mammogrammet med varierende tærskler. For at give et større billedbillede indeholder for -løkken koden fra trin 3 til trin 7. Så hvert binært billede vil blive analyseret for abnormiteter. Derudover er dette for loop indkapslet i et andet for loop, der importerer et nyt mammogrambillede fra databasen i hver iteration.
Trin 4: Find abnormiteter for hvert binært billede
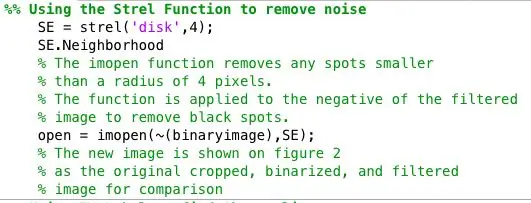
Jeg bearbejdede yderligere de binære billeder ved hjælp af strel -funktionen i forbindelse med imopen for at fjerne baggrundsstøj. Det binære billede fra det foregående trin inverteres og filtreres ved hjælp af kvarteret defineret af SE. Derefter brugte jeg bwlabel til at mærke alle områder, der havde mindst 8 forbundne pixels.
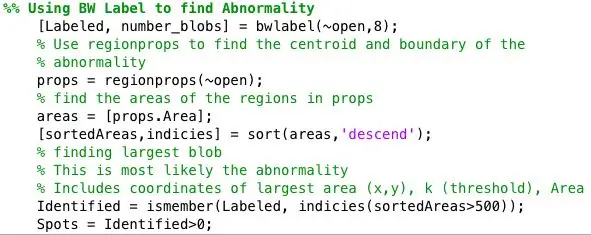
Region -rekvisitfunktionen blev brugt til at finde centroid- og arealeegenskaberne for hvert sted identificeret ved bwlabel.
Derefter blev alle pletter større end 500 pixels identificeret ved hjælp af ismember. Centroiderne for de identificerede pletter blev afbildet på et billede, der kun viste pletter større i areal end 500. Area Identified = ismember (Mærket, indikerer (sorterede områder> 500)); Pletter = Identificeret> 0;
Trin 5: Planlægning af den diagnosticerede masseplacering og -størrelse til visuel sammenligning
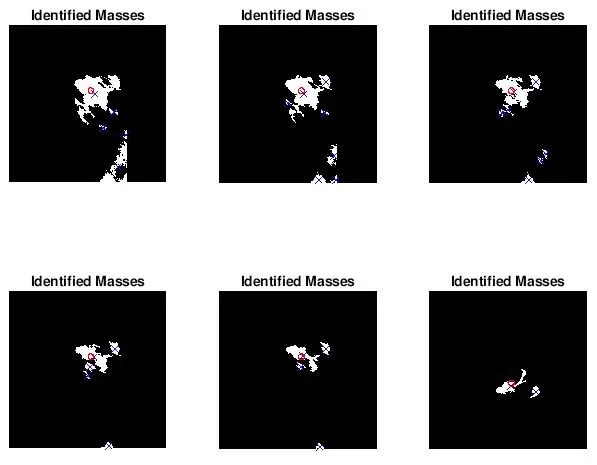

Jeg ville se, om pletterne fundet af bwlabel var korrekte. Jeg gjorde dette på to måder. Jeg analyserede først nøjagtigheden af min klassifikator ved at foretage en visuel sammenligning. Jeg plottede simpelthen den faktiske størrelse og placeringen af abnormiteten (rød cirkel) og placeringen bestemt af koden (blå x) på det forbehandlede mammogrambillede. De seks billeder ovenfor viser virkningerne af at øge tærskelværdien for gråtoner.
Trin 6: Implementering af den anden sammenligningsmetode

Den anden måde, hvorpå jeg testede klassifikatoren og tærskelværdierne, var ved at bestemme, om de placeringer, klassifikatoren fandt, var inden for en vis afstand fra de diagnosticerede abnormitetskoordinater. Jeg gemte de tærskler, for hvilke mindst et af de identificerede punkter var inden for 1,5*r fra den kendte abnormitet til en separat tekstfil kaldet Mammogram Data. Formålet med dette var at finde den minimumstærskel, der er nødvendig for, at min klassifikator kan identificere abnormiteten.
Trin 7: Analyse af indsamlede data
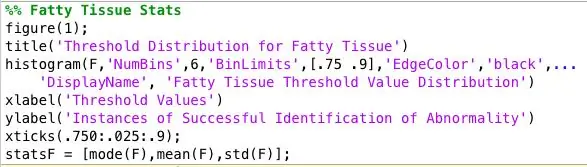
Jeg kørte programmet på alle de unormale mammogrambilleder, og jeg stod tilbage med en enorm tekstfil med data. For at finde den bedste tærskel for hver type væv organiserede jeg dataene efter vævstype og afbildede et histogram over tærskelværdierne for hver vævstype. Den korrekte tærskelværdi blev besluttet på hvilken tærskel, der gav de mest nøjagtige resultater for hver vævstype. Jeg gemte disse data for at uploade til min klassifikator.
Trin 8: Lav din egen klassificering

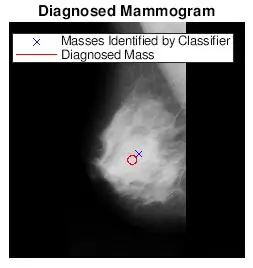
Efter at jeg fandt de mest passende tærskelværdier for hver vævstype, redigerede jeg min originale kode for at få en bruger til at indtaste billednummeret og vævstypen for at vælge tærsklen for mammogrambilledet. Jeg plottede derefter den diagnosticerede mammogramplacering med de fundne placeringer på de originale mammogrambilleder. Jeg ville gøre det sjovere, så jeg programmerede en funktion til at beskære et cirkulært område omkring ROI. Brugeren vil blive instrueret i at vælge et midtpunkt og flere punkter, der bedst omfatter ROI. Jeg vedhæfter begge matlab -filer her.
Trin 9: Forbedringer? Nogen tanker?
Da jeg skrev dette instruerbare, begyndte jeg at se mange forbedringer, jeg kunne foretage i klassifikatoren, såsom at finde måder at skelne mellem forskellige typer masser identificeret baseret på teksturanalyse eller forbedre min test for nøjagtighedsafsnit af SandBoxProject. fil. Da dette var et projekt med en deadline, måtte jeg stoppe et eller andet sted, men jeg håber, at jeg kan bruge billedbehandlingsfærdighederne, jeg lærte i andre applikationer. Jeg vedhæftede også den fil, der blev brugt til batchbehandling af alle de unormale mammogrambilleder.
Anbefalede:
Billig NMEA/AIS Hub - RS232 til Wifi Bridge til brug ombord: 6 trin

Billig NMEA /AIS Hub - RS232 til Wifi Bridge til brug ombord: Opdater 9. januar 2021 - Tilføjet ekstra TCP -forbindelse og genbrug sidste forbindelse, hvis flere klienter opretter Opdatering 13. december 2020 - Tilføjet ingen konfigurationsversion af koden til både med eksisterende routere Introduktion Denne NMEA / AIS RS232 til WiFi -bro er
I2C / IIC LCD -skærm - Brug et SPI LCD til I2C LCD Display Brug SPI til IIC modul med Arduino: 5 trin

I2C / IIC LCD -skærm | Brug en SPI LCD til I2C LCD -skærmen Brug af SPI til IIC -modulet med Arduino: Hej fyre, da en normal SPI LCD 1602 har for mange ledninger at tilslutte, så det er meget svært at grænseflade det med arduino, men der er et modul på markedet, som kan konverter SPI -skærm til IIC -skærm, så du skal kun tilslutte 4 ledninger
Brug 1 analog indgang til 6 knapper til Arduino: 6 trin
Brug 1 analog indgang til 6 knapper til Arduino: Jeg har ofte spekuleret på, hvordan jeg kunne få flere digitale indgange til min Arduino. Det gik for nylig op for mig, at jeg skulle kunne bruge en af de analoge indgange til at indføre flere digitale input. Jeg lavede en hurtig søgning og fandt ud af, hvor folk var
Orange PI HowTo: Konfigurer den til brug sammen med bilens bagskærm og HDMI til RCA -adapter: 15 trin

Orange PI Sådan gør du det: Brug det med bilens bagskærm og HDMI til RCA -adapter: FOREWORD. Det ser ud til, at hver anden bruger et stort og endnu større tv -apparat eller en skærm med et fjollet Orange PI -kort. Og det ligner lidt overkill, når det er beregnet til integrerede systemer. Her har vi brug for noget lille og noget billigt. Som en
Brug af Parallax Basic Stamp II til at ringe til en dørklokke eksternt: 4 trin (med billeder)

Brug af Parallax Basic Stamp II til at ringe til en dørklokke eksternt: Problemet? En hund der bliver alt for begejstret når dørklokken ringer. Løsningen? Ring på døren på tilfældige tidspunkter, hvor ingen er der, og ingen svarer på den for at modvirke hunden - for at bryde foreningen, som en ringende dørklokke e
