Indholdsfortegnelse:
- Trin 1: Vælg og konfigurer billede til analyse
- Trin 2: Tærskel og GUI
- Trin 3: Plotte konturer og celledistribution
- Trin 4: Konverter cellebillede
- Trin 5: Tæl celler og beregn cellekonfluens
- Trin 6: Cellernes rundhed
Video: BME 60B Sandbox Project: 6 trin

2024 Forfatter: John Day | [email protected]. Sidst ændret: 2024-01-30 08:26
Vores Sandbox -projekt har til formål at hjælpe forskere inden for det biologiske felt med at analysere celleprøver og finde ud af betingelserne for deres celler. Efter at brugeren har indtastet et billede af deres celleprøve, behandler vores kode billedet for at gøre det klar til celletælling ved at konvertere billedet til gråtoner og binært. Koden anvender tærskelværdi til at fjerne overskydende information, der ikke vedrører de faktiske celler for nøjagtigt at finde cellemasserne. Efter at have talt antallet af celler i billedet, giver vores kode billedets pixelstørrelse og finder derefter fordelingen af celleområdet ved at plotte antallet af celler versus cellearealet for at give brugeren sammenløbet, som er procentdel af overfladen af en kulturskål, der er dækket af cellerne af interesse. Baseret på sammenløbet kan forskeren afgøre, om cellerne skal passeres eller ej; passage af celler refererer til at udvide liv eller antal celler eller mikroorganismer ved at overføre noget af kulturen til et frisk vækstmedium, og det er nyttigt, når celler vokser for store eller løber tør for mad. Efter at have kørt koden og behandlet billedet, kan brugeren beslutte, om de accepterer eller afviser resultaterne, og fortsætter med at justere tærsklen for bedre hentning af data, hvis det er nødvendigt.
Trin 1: Vælg og konfigurer billede til analyse
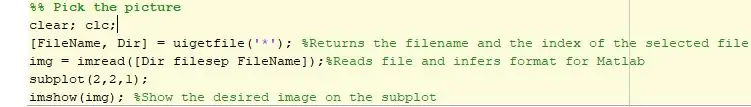
Det første trin i vores kode er at vælge det passende billede og konfigurere det til analyse i Matlab. Vi er i stand til at vælge billedet ved hjælp af uigetfile -funktionen, som giver os mulighed for at få fat i enhver fil og indsætte den i programmet. Derefter læser vi billedet ved hjælp af imread og får det konfigureret til analyse i Matlab. Det valgte billede vises derefter på en underplot.
Trin 2: Tærskel og GUI
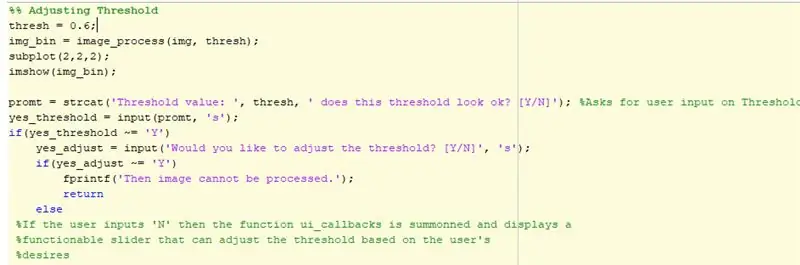
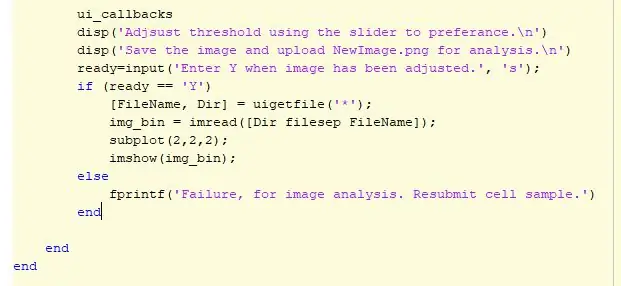
I begyndelsen af koden vælges et billede ved hjælp af "uigetfile" og derefter definerer dette billede med en variabel. Variablen vil derefter blive brugt til at identificere koden, når der køres forskellige analyser. En 2x2 subplot er oprettet i figur. I position 1 vises det originale billede. Det næste afsnit af koden er, hvor justering af tærskler finder sted. Oprindeligt bruges en standard på en tærskel på 0,6 og vises i position 2 på delplottet. En if-sætning bruges derefter til at afgøre, om brugeren vil beholde tærsklen eller justere den. Brugeren kan end justere tærsklen ved hjælp af en GUI, der indeholder billedet ved forskellige tærskelbetingelser, skyderen og en gem -knap. Når tærsklen er indstillet, klikker brugeren på knappen Gem for at gemme billedet, og det vil blive gemt i brugernes MATLAB -filer som en-p.webp
Trin 3: Plotte konturer og celledistribution
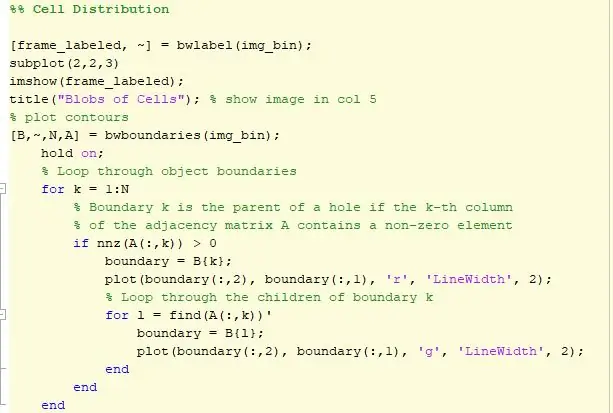
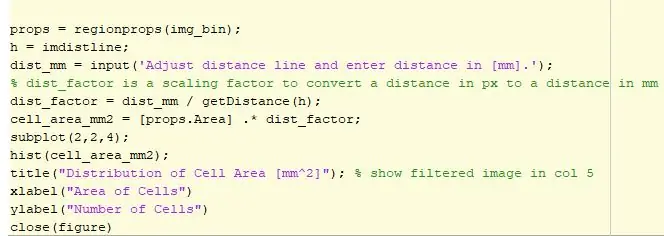
Den næste del af koden tegner konturerne. Der er en sløjfe, hvor cellerne er omsluttet af en rød omkreds, og de celler, der er på toppen af en anden celle, er skitseret med grønt. Det skitserede billede vises derefter i position 3 med en interaktiv afstandslinje. Denne linje vil bestemme antallet af pixels i linjen justeret af brugeren til en pixel til millimeter konverter. Afstandsfaktoren multipliceres derefter med det område, der er bestemt af regionens rekvisitter, og området udtrykkes nu i millimeter i kvadrat. Dataene plottes derefter ved hjælp af et histogram for at se fordelingen af celler efter sit område. Dette histogram vises derefter i position 4.
Trin 4: Konverter cellebillede

I dette trin tog vi det gråtonede billede og binærede, filtrerede og vendte det. Ved at udføre disse funktioner på billedet fjernes støjende pixels, der kan forveksles med celler og gjorde billedet mere glat og blødt omkring cellens kanter. Dette blev gjort for at udtrække cellerne i billedet som adskillelige "klatter", der adskilte sig i intensitet fra baggrunden. "Blobs" var hvide billeder med høj intensitet, og baggrunden var sort. Hvis det var tilladt lidt mere tid, ville vi have brugt en anden Blob -billedkonvertering i stedet for funktionen imbinarize for at være mere præcis og mere passende til vores billeder, men vi havde brug for mere tid til at undersøge og implementere funktionen.
Trin 5: Tæl celler og beregn cellekonfluens

I dette trin i koden havde vi til formål at tælle antallet af celler, der var i billedet. Vi brugte primært funktionen regionprops til at beregne områderne af klatterne, og hvis området blev beregnet til at være i vores ønskede grænse, ville det blive afbildet på en delplot. Grænserne blev sat for at fjerne små støjende pixels eller store intensiteter, der ikke var celler. Celltælleren ville derefter tælle de centroider, der derefter blev afbildet og tilføje dem til tælleren i for -løkken. Når cellernes områder var bestemt, var vi i stand til at beregne konfluens. Hovedvigtigheden af dette trin i koden var at finde cellernes sammenløb, og dette var afgørende for vores slutmål med koden. Vi gjorde dette ved at beregne ved at opsummere pixels (sum (allAreas)) i hver klat og derefter dividere det med billedets samlede pixelværdi (numel (img)). Dette forhold ville give os sammenløbet, og hvis det blev bestemt til at være større end 80%, end det er tid for cellerne at blive passeret af forskeren. Vi stræbte efter at være så præcise og præcise som muligt, men med den begrænsede tid opstod der unøjagtighed. Hvis tiden tillod det, ville vi have kigget på måder at gøre tællingen af klatter mere præcise, såsom flere filtreringsteknikker og/eller en Hough Transform, da der ikke var gjort nok forskning endnu for at prøve den filtreringsteknik
Trin 6: Cellernes rundhed
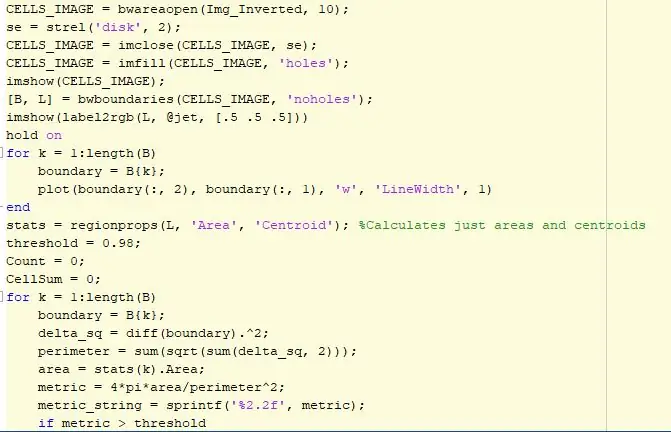

Inden vi kan måle rundhed af klat i et billede, skal vi konvertere fra RGB til gråtoner, binarisere, vende og filtrere billedet. En filtreringsteknik bruger funktionen bwareaopen, som filtrerer billedet af interesse, og det fjerner eventuelle specifikationer eller pixels, der er for små, der ikke repræsenterer størrelsen på en celle. Et strukturelement oprettes med en form på en disk og et område på 2, og det bruges til at udfylde eventuelle huller i baggrunden eller i cellerne. Derefter bruger vi funktionen bwboundaries, som sporer blobs og og gemmer det i en matrix. Vi mærker derefter billedet ved hjælp af forskellige farver, så det bliver et klarere visuelt. Ved siden af en for -loop, der kører i henhold til antallet af objekter og huller, der findes i billedet, sætter den en grænse omkring klatterne, der svarer til dette array. Når denne sløjfe er færdig, begynder en anden sløjfe igen efter antallet af objekter og huller, der findes i billedet. Denne gang bruger vi funktionen regionprops, der samler bestemte egenskaber som område fra arrayet og gemmer informationen, der indeholder antallet af objekter og huller. Ved hjælp af disse oplysninger beregner vi arealet og omkredsen af disse objekter ved hjælp af centroidformen. En tærskel er sat for at sammenligne resultaterne, når vi har beregnet metriske enheden for et cirkulært objekt og tegner denne måling som tekst ved siden af de beregnede centroider. Den endelige kode vil vise rundheden af de forskellige celler, der findes i billedet, og alle værdier tæt på den værdi, man vil have, er mere runde end andre. En værdi på 1 indebærer, at cellen er perfekt rund og er i god stand til at passeres.
Anbefalede:
Gimbal Stabilizer Project: 9 trin (med billeder)

Gimbal Stabilizer Project: Sådan laver du en GimbalLær hvordan du laver en 2-akset gimbal til dit actionkamera I dagens kultur elsker vi alle at optage video og fange øjeblikke, især når du er en indholdsskaber som mig, har du sikkert stået over for spørgsmålet om sådan rystende vide
Automatiseret EKG-BME 305 Slutprojekt Ekstra kredit: 7 trin

Automatiseret EKG-BME 305 Slutprojekt Ekstra kredit: Et elektrokardiogram (EKG eller EKG) bruges til at måle de elektriske signaler, der produceres af et bankende hjerte, og det spiller en stor rolle i diagnosen og prognosen for hjerte-kar-sygdomme. Nogle af de oplysninger, der er opnået fra et EKG, omfatter rytmen
Automatiseret Pet-Food Bowl Project: 13 trin

Automated Pet-Food Bowl Project: Denne instruktive vil skildre og forklare, hvordan man bygger en automatiseret, programmerbar dyrefoder med vedhæftede madskåle. Jeg har vedhæftet en video her, der viser, hvordan produkterne fungerer, og hvordan det ser ud
Akustisk levitation med Arduino Uno trin for trin (8 trin): 8 trin
Akustisk levitation med Arduino Uno Step-by Step (8-trin): ultralyds lydtransducere L298N Dc kvindelig adapter strømforsyning med en han-DC-pin Arduino UNOBreadboard Sådan fungerer det: Først uploader du kode til Arduino Uno (det er en mikrokontroller udstyret med digital og analoge porte til konvertering af kode (C ++)
BME 305 EEG: 4 trin

BME 305 EEG: Et elektroencefalogram (EEG) er en enhed, der bruges til at måle den subjektive elektriske hjerneaktivitet. Disse tests kan være meget nyttige til at diagnosticere forskellige hjernesygdomme. Når du prøver at lave et EEG, er der forskellige parametre, der ikke
